Biogenèse des ARNs et homéostasie du génome
Benoit PALANCADE
Garantir l’intégrité du génome tout en permettant l’expression des gènes est un enjeu caractéristique des cellules vivantes. A ce titre, la production régulée d’ARN messagers est critique pour l’établissement des profils d’expression génétique, mais peut entrer en compétition avec la maintenance ou la duplication des chromosomes. C’est dans ce cadre que notre équipe explore les mécanismes qui régulent les différentes étapes du métabolisme des ARNm, depuis leur synthèse dans le noyau jusqu’à leur export et leur traduction dans le cytoplasme.
Mots-clés : ARN messager ; expression des gènes ; transcription ; stabilité génétique ; hybrides ADN:ARN et R-loops ; traduction localisée ; sumoylation ; pores nucléaires.
+33 (0)157278039 benoit.palancade(at)ijm.fr @PalancadeLab
Garantir l’intégrité du génome tout en permettant l’expression des gènes est un enjeu caractéristique des cellules vivantes. A ce titre, la production régulée d’ARN messagers est critique pour l’établissement des profils d’expression génétique, mais peut entrer en compétition avec la maintenance ou la duplication des chromosomes. C’est dans ce cadre que notre équipe explore les mécanismes qui régulent les différentes étapes du métabolisme des ARNm, depuis leur synthèse dans le noyau jusqu’à leur export et leur traduction dans le cytoplasme, en cherchant notamment à comprendre :
– comment la biogenèse des ARNm est synchronisée avec les autres transactions moléculaires ciblant l’ADN, en s’opposant notamment à la formation d’hybrides ADN:ARNm (ou R-loops) génotoxiques;
– comment ces processus sont organisés dans l’espace, en particulier vis-à-vis des pores nucléaires, les complexes macromoléculaires assurant le transit des ARNm hors du noyau.
La démarche intégrative adoptée par notre équipe pour aborder ces questions combine des approches systématiques et mécanistiques chez un modèle eucaryote unicellulaire, la levure S. cerevisiae.
La synchronisation des différentes activités qui ciblent le matériel génétique dans le noyau est essentielle au maintien de l’homéostasie du génome. Dans ce cadre, le métabolisme des ARNm est récemment apparu comme un facteur déterminant, non seulement pour l’expression des gènes, mais aussi pour le contrôle de la stabilité génétique (Fig. 1). D’une part, la combinatoire d’association des protéines de liaison aux ARN (ou RBP, RNA-binding proteins) contrôle la synthèse des transcrits, leur maturation, leur transport et leur dégradation, permettant ainsi d’établir leur destinée cellulaire, et au final, leur traduction en protéines. Par ailleurs, la biogenèse des ARNm s’oppose à la formation des « R-loops »: ces structures, qui comprennent un hybride ADN:ARNm et un simple brin d’ADN déplacé, peuvent augmenter la sensibilité du matériel génétique aux agents génotoxiques, et perturber sa duplication, représentant ainsi une source d’instabilité génétique chez de nombreuses espèces. Pour contrôler précisément la biogenèse des ARNm, les activités de plusieurs complexes multiprotéiques sont couplées et régulées depuis la synthèse des transcrits jusqu’à leur export hors du noyau et leur traduction. A l’inverse, le découplage de ces différentes étapes, tel qu’observé dans de multiples situations physiologiques, mutantes ou pathologiques (par exemple, dans certains cancers), présente de graves conséquences pour l’expression des gènes et la stabilité du génome (Fig. 1).
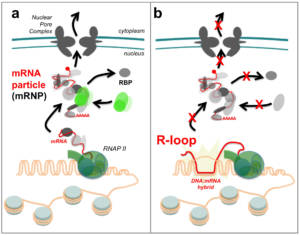
Figure 1 – La biogenèse des particules d’ARNm (mRNPs) et son impact sur l’expression des gènes et la stabilité du génome. a, Synchronisation des différentes étapes de la biogenèse des ARNm : le recrutement de protéines de liaison aux ARNs (RNA-binding protein, RBP) spécifie la destinée cellulaire des transcrits, et s’oppose à la formation des R-loops. b, Découplage des différentes étapes de la biogenèse des ARNm : des défauts de maturation ou d’export des ARNm entrainent la formation de R-loops génotoxiques. RNAP II, RNA polymerase II. Copyright CNRS / IJM / B. PALANCADE.
Les activités de recherche de l’équipe visent à identifier les mécanismes régulateurs qui affectent le métabolisme des ARNs en cis et en trans, et à comprendre leur impact sur l’expression et l’intégrité du génome. Nous nous intéressons notamment à l’influence de la biogenèse des ARNm sur la formation des R-loops et à ses conséquences sur le maintien de la structure et de la stabilité du génome (voir ci-dessous, axe 1), et à l’organisation de ces processus dans l’espace cellulaire, notamment vis-à-vis des pores nucléaires, des complexes macromoléculaires notamment impliqués dans le transport des ARNm vers le cytoplasme (voir ci-dessous, axe 2). Dans ce but, des approches systématiques – génomiques et protéomiques – sont combinées avec des études mécanistiques in vitro et in vivo, principalement chez la levure S. cerevisiae, un système modèle pour comprendre ces processus conservés (Fig. 2).
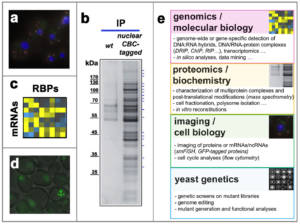
Figure 2 – Biogenèse des ARN et homéostasie du génome : méthodes d’étude. a, La localisation subcellulaire d’ARNm – ici, ceux codant pour des sous-unités des pores nucléaires – est analysée dans des cellules de levure par smFISH (single molecule fluorescence in situ hybridization). b, La composition des particules d’ARNm est analysée par spectrométrie de masse après purification à l’aide d’une version étiquetée du complexe de liaison à la coiffe (Cap-binding complex, CBC ; coloration à l’argent après électrophorèse dénaturante). c, L’association des RBPs aux ARNm est analysée à l’échelle du transcriptome en compilant des données d’interaction existantes. d, La formation de foyers de réparation (marqués par une version fluorescente de Rad52) est détectée par imagerie sur des levures mutantes pour un facteur de biogenèse des ARN (complexe THO), révélant l’instabilité génétique typique de ce mutant. e, Aperçu des différentes approches utilisées dans l’équipe. Copyright CNRS / IJM / B. PALANCADE.
AXE 1 – BIOGENÈSE DES ARNM ET MAINTIEN DE L’INTÉGRITÉ GÉNÉTIQUE : LES R-LOOPS SOUS CONTRÔLE
Si la formation de R-loops s’est récemment révélée comme largement répandue dans le monde vivant et associée à de multiples processus génomiques, notre compréhension des mécanismes cellulaires qui modulent leur accumulation reste encore limitée. Pour identifier les facteurs qui contrôlent en cis et en trans l’apparition de ces structures, nous avons combiné des approches génomiques et génétiques chez la levure, mettant ainsi en évidence de nouveaux aspects de leur régulation.
1.1. Un nouveau rôle pour les introns : protéger les génomes eucaryotes de l’instabilité génétique associée aux R-loops
Le rôle central de l’épissage des introns dans le contrôle de l’expression des génomes nous a amené à évaluer son impact sur la biogenèse des ARNm et le maintien de la stabilité génétique. Pour ceci, nous avons réalisé un crible basé sur un système rapporteur permettant d’identifier les facteurs spécifiques à la biogenèse et à l’export des ARNm issus de gènes comprenant un intron (Bonnet et al., 2015; voir aussi Bonnet & Palancade, 2015). Cette étude a révélé que la présence d’introns atténuait l’effet délétère de mutations associées à l’accumulation de R-loops, suggérant que ces séquences pourraient s’opposer à leur génotoxicité. Pour étendre ces observations à l’echelle du génome, nous avons exploré des données de distribution des R-loops et des dommages à l’ADN chez la levure S. cerevisiae et l’humain, et montré que ces lésions génétiques s’accumulaient préférentiellement sur les gènes dépourvus d’introns (collaboration avec Sergio de ALMEIDA, Université de Lisbonne, Portugal). En modifiant directement le contenu en introns de gènes modèles par des approches d’édition du génome chez la levure, nous avons ensuite établi que l’élimination d’introns endogènes entrainait l’apparition de R-loops, alors que l’insertion d’introns dans des gènes n’en comprenant pas naturellement atténuait la formation de ces structures ainsi que des dommages à l’ADN associés. L’insertion en place d’un intron de différentes séquences capables de s’épisser et/ou de s’associer à des protéines a finalement révélé que c’est la liaison de protéines sur l’ARN, et non le processus d’épissage, qui s’oppose stériquement à la formation des hybrides ADN:ARN. Cette nouvelle fonction des introns dans la protection contre des structures génotoxiques pourrait contribuer à expliquer leur conservation au cours de l’évolution (Fig. 3; Bonnet et al., 2017; Palancade, 2018).
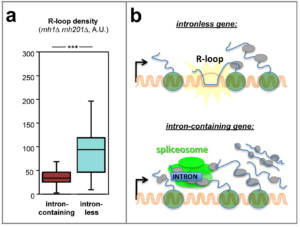
Figure 3 – Les introns protègent les génomes de l’accumulation de R-loops. a, Détection d’un niveau réduit d’hybrides ADN:ARN sur les gènes à introns de la levure S. cerevisiae dans des études génomiques (analyse réalisée dans un mutant des deux RNases H prévenant naturellement l’accumulation d’hybrides). b, Modèle : sur les gènes à introns, le recrutement de la machinerie d’épissage (spliceosome) s’oppose physiquement à la formation des hybrides ADN:ARN. D’après Bonnet et al, 2017. Copyright CNRS / IJM / B. PALANCADE.
1.2. Vers une approche systématique des voies cellulaires contrôlant la formation et la résolution des R-loops
Malgré l’intérêt croissant de la communauté scientifique pour les mécanismes de formation et de genotoxicité des R-loops, aucune approche systématique n’a permis à ce jour d’identifier de manière exhaustive les facteurs cellulaires contrôlant le métabolisme de ces structures. En collaboration avec l’équipe de Rodney ROTHSTEIN (Columbia University Medical Center, NY, USA), nous avons mis sur pied un crible génétique permettant d’identifier les protéines prévenant la formation des R-loops ou assurant leur résolution, limitant ainsi l’instabilité génétique associée. De manière surprenante, plusieurs des facteurs identifiés sont associés à d’autres processus génomiques, confirmant les relations étroites entre métabolisme de l’ARN et de l’ADN, déjà attestées par l’existence de machineries cellulaires ne discriminant pas strictement ces différents substrats (Palancade & Rothstein, 2021). La caractérisation mécanistique des facteurs ainsi identifiés devrait permettre de comprendre à terme comment la biogenèse des ARNm est synchronisée avec d’autres étapes du métabolisme de l’ADN, notamment réplication et réparation.
AXE 2 – L’ORGANISATION SPATIALE DU MÉTABOLISME DES ARNM: UN RÔLE CENTRAL POUR LES PORES NUCLÉAIRES
2.1. Pores nucléaires et biogenèse des ARNm : une affaire de SUMOylation
Lorsque les particules d’ARNm (mRNPs) s’engagent dans la voie d’export sur la face nucléaire des complexes des pores nucléaires (NPCs), elles se trouvent à proximité d’Ulp1SENP2, une enzyme de déconjugaison de SUMO qui régule les modifications post-traductionnelles par ce petit polypeptide (Palancade & Doye, 2008). Pour déterminer si des régulations par SUMOylation modulent ainsi la biogenèse ou le transport des ARNm, nous avons combiné des approches d’exploration de données (Rouviere et al, 2013 ; Rouviere et al, 2018), de protéomique (Bretes et al, 2014) et de transcriptomique (Bonnet et al, 2021). A l’issue d’un crible protéomique permettant la caractérisation des mRNPs à différents stades de leur biogenèse (collaboration avec Marlene OEFFINGER, IRCM, Montréal, Canada, et avec la plateforme de Protéomique ProteoSeine à l’IJM), nous avons notamment montré que les régulations par SUMO contrôlent le recrutement d’un nombre limité de protéines de liaison à l’ARN, dont les facteurs conservés THO et Hek2hnRNP-K. Dans le cas du complexe THO, un facteur clé de la biogenèse des ARNm, ce recrutement contrôlé par SUMO s’oppose à la dégradation de certains ARNm induits en conditions de stress, assurant la survie dans de telles situations (Fig. 4; Bretes et al., 2014). En revanche, l’association régulée d’Hek2 à ses ARNm cibles permet leur répression traductionnelle (collaboration avec Bertrand COSSON, Université de Paris). De manière frappante, les ARNm codant pour des sous-unités des NPCs sont eux-mêmes les principales cibles de cette régulation, révélant une boucle de rétrocontrôle dans laquelle Ulp1 et Hek2 sont respectivement le senseur et l’effecteur d’un mécanisme maintenant l’intégrité des pores nucléaires (Fig. 5; Rouviere et al, 2018). Au delà de ces régulations post-transcriptionnelles, nos études transcriptomiques ont également révélé que les mécanismes de (dé)SUMOylation associés aux pores nucléaires pouvaient contrôler la synthèse d’autres classes d’ARNm (Bonnet et al, 2015), en réprimant notamment la transcription d’éléments transposables (collaboration avec Pascale LESAGE, Hôpital St-Louis, Paris ; Bonnet et al., 2021).
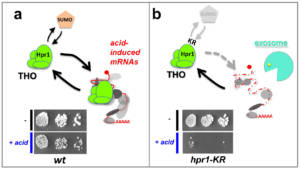
Figure 4 – La SUMOylation régule la biogenèse et la stabilité d’ARNm induits par le stress. a, Dans des cellules sauvages, la SUMOylation du complexe THO permet son association aux ARNm induits par le stress acide. L’expression de ces ARNm permet ainsi la survie dans ces conditions de stress spécifiques (panel du bas). b, Dans des cellules mutantes dans lesquelles la SUMOylation du complexe THO sur sa sous-unité Hpr1 est abolie (hpr1-KR), les ARNm induits par le stress acide voient leur association au complexe THO diminuée, et sont dégradés par l’exosome, entrainant une perte de viabilité dans ces conditions (panel du bas). D’après Bretes et al. 2014. Copyright CNRS / IJM / B. PALANCADE.
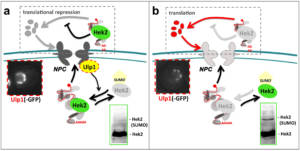
Figure 5 – Une boucle de régulation contrôlant l’intégrité d’un complexe multiprotéique. a, Quand les complexes des pores nucléaires (NPCs) sont correctement assemblés, les niveaux élevés d’Ulp1, l’enzyme de déconjuguaison de SUMO associée aux pores, entrainent la dé-SUMOylation de la protéine Hek2 et sa liaison aux ARNm codant pour des sous-unités des NPCs, réprimant leur traduction. b, Dans des situations où les NPCs sont défectueux, les niveaux réduits d’Ulp1 entrainent l’accumulation d’Hek2 sous forme SUMOylée, prévenant son association à ses ARNm cibles et augmentant la synthèse des sous-unités des pores. D’après Rouviere et al, 2018. Copyright CNRS / IJM / B. PALANCADE.
2.2. Des ARNm traduits aux pores nucléaires
L’identification de régulations traductionnelles ciblant collectivement les ARNm codant pour les sous-unités d’un complexe protéique nous a conduit à étudier les couplages entre synthèse et assemblage des polypeptides. En analysant systématiquement les interactions co-traductionnelles entre sous-unités des NPCs, nous avons ainsi pu mettre en évidence que certains blocs constitutifs des pores s’assemblent de manière ordonnée, durant la traduction de leurs constituants, dans le cytoplasme. En revanche, certains ARNm codant pour des protéines de la face interne des NPCs, ou pour d’autres protéines nucléoplasmiques, sont traduits à proximité des pores (collaboration avec Martine COLLART, Université de Genève, Suisse, et Frédéric DEVAUX, Sorbonne Université, Paris). Cette traduction localisée requiert la reconnaissance de signaux d’adressage nucléaire, dans les polypeptides naissants, par des facteurs de transport, les karyophérines, elle-mêmes associées aux NPCs, et permettrait d’éviter l’accumulation délétère de certaines protéines toxiques ou susceptibles de s’agréger dans le cytoplasme (Lautier et al, 2021). En ce sens, ces évènements de traduction localisée rappellent ceux ciblant les protéines présentant une séquence signal pour l’adressage au réticulum endoplasmique, bien que les machineries associées présentent des origines évolutives distinctes (Fig. 6).
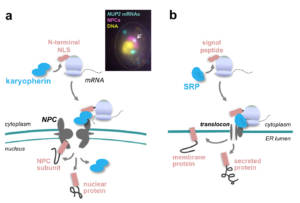
Figure 6 – Des ARNm traduits aux pores nucléaires. a, représentation schématique du mécanisme d’adressage des ARNm traduits aux NPCs, et image typique d’hybridation in situ montrant la localisation de l’ARNm NUP2. b, comparaison avec le mécanisme d’adressage au réticulum endoplasmique (ER) des ARNm codant pour des protéines membranaires ou secrétées. Dans ce dernier cas, la traduction localisée requiert la reconnaissance du peptide signal dans la protéine naissante par la SRP (particule de reconnaissance du signal). Copyright CNRS / IJM / B. PALANCADE.
AUTRES CONTRIBUTIONS DANS LES DOMAINES DE LA BIOGENÈSE DES ARNM ET DE L’HOMÉOSTASIE DU GÉNOME
Dans le cadre des travaux précédemment décrits, nous avons implémenté différentes approches pour étudier le métabolisme des ARNm et leur impact sur la stabilité du génome, lesquelles ont aussi été mises en œuvre dans le cadre de collaborations. Nous avons notamment participé à différentes études en contribuant à la caractérisation fonctionnelle de complexes ribonucléoprotéiques ou de modifications post-traductionnelles par SUMO (Infantino et al., 2019 ; Talhaoui et al., 2018 ; Delaveau et al, 2016), au suivi de la localisation d’ARNs in situ (Andric et al, 2021 ; Kramarz et al, 2020 ; Hazra et al, 2020) ou à des mesures d’instabilité génétique à l’aide de rapporteurs dédiés (Babour et al., 2016; Fritzen et al., 2016).
Membres
 Anna BABOUR, Chercheur, PALANCADE LAB+33 (0)1 57 27 80 39, bureau 354B
Anna BABOUR, Chercheur, PALANCADE LAB+33 (0)1 57 27 80 39, bureau 354B Karine CASIER, PALANCADE LAB+33 (0)1 57 27 80 39, bureau 354B
Karine CASIER, PALANCADE LAB+33 (0)1 57 27 80 39, bureau 354B Coralie GONCALVES PEREIRA PERRUCA, Assistante-ingénieure en biologie, PALANCADE LAB+33 (0)1 57 27 80 38, bureau 354B
Coralie GONCALVES PEREIRA PERRUCA, Assistante-ingénieure en biologie, PALANCADE LAB+33 (0)1 57 27 80 38, bureau 354B Raphael MANGIONE, Doctorant, PALANCADE LAB+33 (0)1 57 27 80 38, bureau 354B
Raphael MANGIONE, Doctorant, PALANCADE LAB+33 (0)1 57 27 80 38, bureau 354B Mohamed MLIH, Ingénieur en biologie, PALANCADE LAB+33 (0)1 57 27 80 38, bureau 354B
Mohamed MLIH, Ingénieur en biologie, PALANCADE LAB+33 (0)1 57 27 80 38, bureau 354B Myriam ZHENG, Doctorante, PALANCADE LAB+33 (0)1 57 27 80 39, bureau 354B
Myriam ZHENG, Doctorante, PALANCADE LAB+33 (0)1 57 27 80 39, bureau 354B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
Penzo, A., Dubarry, M., Brocas, C., Zheng, M., Mangione, R. M., Rougemaille, M., Goncalves, C., Lautier, O., Libri, D., Simon, M.-N., Géli, V., Dubrana, K., & Palancade, B. (2023).
A R-loop sensing pathway mediates the relocation of transcribed genes to nuclear pore complexes.
Nature Communications, 14(1), 5606. https://doi.org/10.1038/s41467-023-41345-z
Bonnet A, Chaput C, Palmic N, Palancade B* & Lesage P* (2021)
A nuclear pore sub-complex restricts the propagation of Ty retrotransposons by limiting their transcription.
PLoS Genet. 17(11):e1009889
https://journals.plos.org/plosgenetics/article?id=10.1371/journal.pgen.1009889
Palancade, B & Rothstein, R (2021)
The ultimate (mis)-match: when DNA meets RNA.
Cells 10(6):1433
https://www.mdpi.com/2073-4409/10/6/1433
Lautier O, Penzo A, Rouvière JO, Chevreux G, Collet L, Loïodice I, Taddei A, Devaux F, Collart MA, Palancade B. (2021)
Co-translational assembly and localized translation of nucleoporins in nuclear pore complex biogenesis.
Mol Cell. 2021 Apr 4:S1097-2765(21)00225-2.
https://www.sciencedirect.com/science/article/abs/pii/S1097276521002252
Image de couverture: https://www.cell.com/molecular-cell/issue?pii=S1097-2765(20)X0012-8
Voir les actualités du CNRS (INSB) et de l’Université de Paris.
https://insb.cnrs.fr/fr/cnrsinfo/des-arn-messagers-traduits-aux-pores-nucleaires
https://u-paris.fr/des-proteines-cellulaires-fabriquees-a-la-surface-du-noyau/
Rouviere JO, Bulfoni M, Tuck A, Cosson B, Devaux F & Palancade B. (2018)
A SUMO-dependent feedback loop senses and controls the biogenesis of nuclear pore subunits.
Nature communications 9(1):1665
https://www.nature.com/articles/s41467-018-03673-3
Bonnet A, Grosso AR, Elkaoutari A, Coleno E, Presle A, Sridhara SC, Janbon G, Géli V, de Almeida SF & Palancade B. (2017)
Introns protect eukaryotic genomes from transcription-associated genetic instability
Mol Cell 67(4):608-621.e6
https://www.sciencedirect.com/science/article/pii/S1097276517304963?via%3Dihub
Voir le commentaire dans Science et l’actualité du CNRS(INSB):
http://science.sciencemag.org/content/357/6354/883.5
https://www.cnrs.fr/insb/recherche/parutions/articles2017/b-palancade.html
Cet article a été élu “article de l’année 2017” par la SFBBM (Société Française de Biochimie et de Biologie Moléculaire)
https://www.sfbbm.fr/index.php?option=com_content&view=featured&Itemid=364&lang=fr
Bonnet A, Bretes H & Palancade B.(2015)
Nuclear pores affect distinct stages of intron-containing gene expression
Nucleic Acids Research 43(8):4249-61
https://academic.oup.com/nar/article/43/8/4249/2414571
Bretes H, Rouviere JO, Léger T, Oeffinger M, Devaux F, Doye V and Palancade B. (2014)
Sumoylation of the THO complex regulates the biogenesis of a subset of mRNPs
Nucleic Acids Research 42(8):5043-58
https://academic.oup.com/nar/article/42/8/5043/1069049
Bonnet A & Palancade B. (2014)
Regulation of mRNA trafficking by nuclear pore complexes
Genes 5 :767-791 Special issue « Mechanisms of mRNA export »
http://www.mdpi.com/2073-4425/5/3/767/htm
Rouviere JO, Geoffroy MC and Palancade B. (2013)
Multiple cross talks between mRNP biogenesis and SUMO
Chromosoma 122(5):387-99
https://link.springer.com/article/10.1007%2Fs00412-013-0408-y
Publications
Revue
Raphaël Mangione (depuis Octobre 2020), Université de Paris. Caractérisation fonctionnelle de nouveaux facteurs contrôlant la dynamique des R-loops, une source d’instabilité génétique associée à la transcription
Arianna Penzo (depuis Octobre 2018), Université de Paris. Exploring novel mechanisms controlling R-loop-dependent genetic instability
Jérome Rouvière (2012-2016), Université Paris XI. Etude du rôle de la SUMOylation dans le métabolisme des ribonucléoparticules d’ARNm (mRNPs)
Hugo Bretes Rodrigues (2008-2012), Université Paris XI. Etude de la régulation du métabolisme des ARN messagers chez la levure Saccharomyces cerevisiae.
Collaborations en cours
Andres AGUILERA, CABIMER, Sevilla, Spain
Sergio de ALMEIDA, Instituto de Medicina Molecular, University of Lisboa, Portugal
Frédéric DEVAUX & Mathilde GARCIA, Sorbonne Université, Paris, France
Karine DUBRANA, Commissariat à l’Energie Atomique, Fontenay-aux-Roses, France
Gilles FISCHER, Sorbonne Université, Paris, France
Pierre-Henri GAILLARD, Centre de Recherche en Cancérologie de Marseille, France
Vincent GELI, Centre de Recherche en Cancérologie de Marseille, France
Jean-René HUYNH & Anahi MOLLA-HERMAN, Collège de France, Paris, France
Pascale LESAGE, Hôpital St-Louis, Paris, France
Domenico LIBRI, Institut Jacques Monod, Paris, France
Rodney ROTHSTEIN, Columbia University Medical Center, New York, USA
Mathieu ROUGEMAILLE, I2BC, Gif s/Yvette, France
Collaborations passées
Martine COLLART, Faculté de Médecine, Université de Genève, Switzerland
Bertrand COSSON, Epigenetics, Université de Paris, France
Guilhem JANBON, Institut Pasteur, Paris, France
Gerard MAZON, Institut Gustave Roussy, Villejuif, France
Marlene OEFFINGER, IRCM, Montréal, Canada
Sébastien STORCK, Hôpital Necker, Paris, France
Françoise STUTZ, Université de Genève (Sciences III), Switzerland
Les recherches de l’équipe sont soutenues par les établissements publics, agences de financement et associations caritatives suivantes :


