Oncologie moléculaire et pathologies ovariennes
Reiner A. VEITIA
La thématique de l’équipe est l’étude du développement et du fonctionnement de l’ovaire par le biais de la génétique moléculaire. Cette recherche s’effectue via l’analyse moléculaire de différentes pathologies telles que le syndrome du blépharophimosis, l’insuffisance ovarienne prématurée (IOP) ou les tumeurs des cellules de la granulosa (GCT), un des types de cellules somatiques de l’ovaire. Toutes ces maladies impliquent des altérations du facteur de transcription (FT) FOXL2. Au fil des ans, nous avons contribué à une meilleure compréhension de sa fonction moléculaire, de ses mécanismes pathogènes, de ses cibles et de ses partenaires. Nous utilisons FOXL2 comme modèle pour explorer les fondements de la reconnaissance d’une cible spécifique par un FT et l’influence des partenaires et des modifications post-traductionnelles dans ce processus. Récemment nous avons signalé la présence d’insertions dans l’oncogène AKT1 probablement responsables des GCT de forme juvénile, ouvrant la voie à de nouvelles recherches au sein de l’équipe. Enfin, nous recherchons par séquençage d’exomes de cas familiaux et isolés d’IOP de nouveaux gènes incriminés dans cette pathologie. Nous avons ainsi montré l’implication dans les IOP de protéines tels que la cohésine STAG3 ou plus récemment MEIOB.
Mots-clés : Ovaire, facteur de transcription, régulation géniques, analyse omiques, IOP, cancer
+33 (0)157278116 reiner.veitia(at)ijm.fr
L’objectif principal de la recherche effectuée par notre équipe est l’étude de la fonction ovarienne à travers la génétique moléculaire. Un sujet historique du groupe a été l’analyse du syndrome du Blépharophimosis (anomalies craniofaciales et une insuffisance ovarienne prématurée ou IOP), en raison d’altérations du facteur de transcription FOXL2. Au fil des années, nous avons contribué à une meilleure compréhension de la fonction moléculaire de ce dernier, de ses mécanismes de pathogénicité et à la découverte de nombreuses cibles et partenaires. FOXL2 fournit un excellent modèle pour étudier la régulation combinatoire de la transcription, car il module une série de processus apparemment indépendants (p.ex. le métabolisme du cholestérol et des stéroïdes, l’apoptose, le cycle cellulaire, la détoxification des radicaux libres, etc.). Ainsi, nous utilisons FOXL2 comme modèle pour explorer les bases de la reconnaissance spécifique des cibles par un facteur de transcription et l’influence des partenaires et des modifications post-traductionnelles sur ce processus. Pour ce faire nous utilisons les outils de pointe de la génomique et le protéomique.
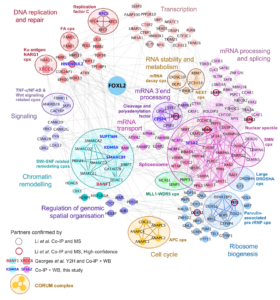
Légende. Interactome de FOXL2 : réseau d’interactions protéine-protéine pour l’ensemble des 255 partenaires de FOXL2 identifiés par spectrométrie de masse.
FOXL2 a également ouvert une voie de recherche concernant les tumeurs des cellules de la granulosa ovarienne (GCT), qui représentent jusqu’à 8% de tous les cancers de l’ovaire. Deux sous-types distincts de GCTs ont été décrits : les formes juvénile et adulte. Le type adulte apparaît le plus souvent pendant la périménopause et se caractérise par des récidives tardives (jusqu’à 40 ans après le traitement de la tumeur primaire). La forme juvénile (JGCT) a tendance à se produire dans la période prépubertaire. Une mutation somatique récurrente dans FOXL2, conduisant à la substitution p.C134W a été détectée dans plus de 97% des AGCT, suggérant que cette mutation est un élément clé dans leur formation. Nous étudions activement les mécanismes pathogéniques de cette mutation en utilisant des lignées cellulaires modifiées par CRISPR-Cas9 et des outils génomiques.
La base moléculaire des JGCT est moins bien comprise. Nous avons récemment trouvé des duplications en tandem dans le cadre de lecture de l’oncogène AKT1 qui affectent le pleckstrin-homology domain (PHD) de la protéine dans plus de 60% des JGCT. Les effets de ces mutations sont actuellement étudiés avec des modèles cellulaires et drosophile.
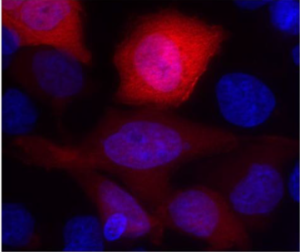
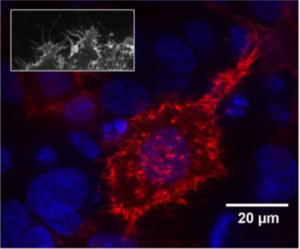
Légende : Cellules HeLa exprimant la version normale d’AKT1 (à droite) et une des versions mutées trouvées dans des tumeurs de l’ovaire (à gauche) (noyau : bleu, AKT1 : rouge).
Enfin, nous profitons de la puissance de la technologie génomique actuelle pour découvrir de nouveaux gènes impliqués dans l’infertilité féminine due à une IOP, en effectuant le séquençage d’exomes dans des cas familiaux et isolés de ce trouble. Les fonctions des gènes candidats découverts lors de ces analyses sont ou seront étudiées en utilisant des approches moléculaires et des modèles animaux.
Membres







Despoina Maria CHOUSIANITI, Erasmus
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
Reply to “An alternative miRISC targets a cancer-associated coding sequence mutation in FOXL2”. Veitia RA, Pilsworth J, Todeschini AL, Huntsman D.EMBO J. 2021 Aug 16;40(16):e107517. doi: 10.15252/embj.2020107517.PMID: 34396573
FOXL2 in adult-type granulosa cell tumour of the ovary: oncogene or tumour suppressor gene? Pilsworth JA, Todeschini AL, Neilson SJ, Cochrane DR, Lai D, Anttonen M, Heikinheimo M, Huntsman DG, Veitia RA.J Pathol. 2021 Nov;255(3):225-231. doi: 10.1002/path.5771. Epub 2021 Sep 1.PMID: 34338304
Insights into the pathogenicity of missense variants in the forkhead domain of FOX proteins underlying Mendelian disorders. Bermúdez-Guzmán L, Veitia RA. Hum Genet. 2021 Jul;140(7):999-1010.
Forkhead Transcription Factors in Health and Disease. Herman L, Todeschini AL, Veitia RA.Trends Genet. 2021 May;37(5):460-475 (Review).
Genomic exploration of the targets of FOXL2 and ESR2 unveils their implication in cell migration, invasion, and adhesion. Herman L, Legois B, Todeschini AL, Veitia RA.FASEB J. 2021 Apr;35(4):e21355. doi: 10.1096/fj.202002444R.PMID: 33749886
A missense in HSF2BP causing primary ovarian insufficiency affects meiotic recombination by its novel interactor C19ORF57/BRME1. Felipe-Medina N, Caburet S, Sánchez-Sáez F, Condezo YB, de Rooij DG, Gómez-H L, Garcia-Valiente R, Todeschini AL, Duque P, Sánchez-Martin MA, Shalev SA, Llano E, Veitia RA, Pendás AM.Elife. 2020 Aug 26;9:e56996. doi: 10.7554/eLife.56996.PMID: 32845237 Free
An exome-wide exploration of cases of primary ovarian insufficiency uncovers novel sequence variants and candidate genes. Alvarez-Mora MI, Todeschini AL, Caburet S, Perets LP, Mila M, Younis JS, Shalev S, Veitia RA.Clin Genet. 2020 Sep;98(3):293-298. doi: 10.1111/cge.13803. Epub 2020 Jul 28.PMID: 32613604
DHH pathogenic variants involved in 46,XY disorders of sex development differentially impact protein self-cleavage and structural conformation. Elzaiat M, Flatters D, Sierra-Díaz DC, Legois B, Laissue P, Veitia RA.Hum Genet. 2020 Nov;139(11):1455-1470.
Conventional and unconventional interactions of the transcription factor FOXL2 uncovered by a proteome-wide analysis. Penrad-Mobayed M, Perrin C, Herman L, Todeschini AL, Nigon F, Cosson B, Caburet S, Veitia RA.FASEB J. 2020 Jan;34(1):571-587. doi: 10.1096/fj.201901573R. Epub 2019 Nov 25.PMID: 31914586
A truncating MEIOB mutation responsible for familial primary ovarian insufficiency abolishes its interaction with its partner SPATA22 and their recruitment to DNA double-strand breaks. Caburet S, Todeschini AL, Petrillo C, Martini E, Farran ND, Legois B, Livera G, Younis JS, Shalev S, Veitia RA.EBioMedicine. 2019 Apr;42:524-531. doi: 10.1016/j.ebiom.2019.03.075. Epub 2019 Apr 15.PMID: 31000419
Publications
Revues
Chapitre de livre
2016-2020 : Laëtitia Herman, doctorat soutenu le 22/09/2020 « Caractérisation moléculaire du rôle de FOXL2 et de ses partenaires dans l’ovaire sain et pathologique »
2012-2015 : Laurianne Bessière, doctorat soutenu le 16/09/2015 « Exploration génomique et fonctionnelle des tumeurs des cellules de la granulosa ovarienne »
2009-2013 : Adrien Georges, doctorat soutenu le 28/06/2013 « FOXL2 : un déterminant de la signalisation dans l’ovaire »
2007-2011 : Bérénice Benayoun, doctorat soutenu le 28/01/2011 « FOXL2 : un facteur de transcription essentiel de l’ovaire, à l’interface entre la réponse au stress cellulaire et la suppression tumorale »
2006-2009 : Aurélie Dipiétromaria, doctorat soutenu le 28/09/2009 « Etudes des conséquences fonctionnelles des mutations de FOXL2 : gène impliqué dans le syndrome du BPES »
2005-2008 : Franck Batista Pelaez, doctorat soutenu le 13/06/2008 « Les cibles transcriptionnelles de FOXL2, un facteur de transcription impliqué dans le développement et le maintien de la fonction ovarienne »
2005-2008 : Lara Moumné, doctorat soutenu le 14/05/2008 « Etude fonctionnelle des mutations affectant FOXL2 : un acteur clé du développement de l’ovaire chez les vertébrés »
2002-2006 : Julie Cocquet doctorat soutenu le //2006 « Evolution, expression et structure de FOXL2, un gène impliqué dans la fertilité féminine »
Etudes sur les tumeurs des cellules de la granulosa ovarienne adultes (AGCTs) et sur le facteur de transcription FOXL2 : Equipe d’E. de Baere, Ghent Belgique. Équipe de N. Binart, Kremlin-Bicêtre, France. F. Poulat, Montpellier, France. Equipe de M. Anttonen, Helsinki, Finlande. Equipe D. Huntsman, Vancouver, Canada. Equipe d’Alberto Pendas, Salamanca, Espagne.
Etudes sur les tumeurs des cellules de la granulosa ovarienne juvéniles (JGCTs) et sur l’oncoprotéine AKT1 : S. Sarnacki et L. Galmiche, Paris. N. Kalfa et C. Sultan, Montpellier. J. Prat et E. d’Angelo, Barcelona, Espagne. Malcolm Donaldson, Glasgow, United Kingdom.
Etudes sur les gènes impliqués dans les insuffisances ovariennes prématurées : Maria Isabel Alvarez-Mora et Montserrat Mila, Barcelona, Espagne. Equipe de Gabriel Livera, Fontenay aux Roses, France. Johnny S. Younis, Safed, Israel. Equipe de Stavit Shalev, Afula, Israel. Equipe d’Alberto Pendas, Salamanca, Espagne.
1) ANR (National Agency for Research) : OVARYPROTECT : Interaction entre la signalisation PKA et TRIM28 dans la maintenance et la pathologie ovarienne
2) GEFLUC (Groupement des Entreprises Françaises dans la Lutte contre le Cancer): Ovarian cancer: FOXL2-C134W impact on cell-cell or cell-ECM adhesion.

