Organisation spatiale de la cellule
NICOLAS MINC
Notre équipe cherche à comprendre les mécanismes fondamentaux de l’organisation cellulaire :
Comment une cellule détermine-t-elle sa forme ?
Comment sent-elle sa forme ou sa taille ?
Comment ces propriétés géométriques contribuent-elles à la division ou la croissance d’une cellule, d’un embryon ou d’un tissu ?
Mots-clés : Division cellulaire, Cytosquelette, Morphogénèse, Développement embryonnaire, Polarité, Paroi, Mécanobiologie, Cytosquelette, Champignons.
+33 (0)157278052 nicolas.minc(at)ijm.fr @MincLab http://www.minclab.fr/
L’organisation spatiale d’une cellule est sous-jacente à de nombreux processus biologiques, depuis la division ou la migration d’une cellule, jusqu’au développement d’un embryon ou l’architecture de nos tissus. Des défauts d’organisation spatiale sont fréquemment associés à des maladies développementales ou à l’émergence de cancers.
Notre équipe cherche à comprendre les mécanismes fondamentaux de l’organisation cellulaire : Comment une cellule détermine-t-elle sa forme ? Comment sent-elle sa forme ou sa taille ? Comment ces propriétés géométriques contribuent-elles à la division ou la croissance d’une cellule, d’un embryon ou d’un tissue ?
Pour répondre à ces questions, nous inventons et intégrons des outils issus de la physique et de l’ingénierie, telles que la microfabrication, l’imagerie quantitative et les modèles mathématiques, avec des méthodes plus traditionnelles de génétique et de biochimie. Notre but est d’obtenir des informations quantitatives et moléculaires sur les processus morphogénétiques et de définir les principes généraux régissant ces processus chez les organismes mono- et multi-cellulaires.
Les cellules ont des formes et des tailles stéréotypées, souvent associées à leur fonction. Les neurones grandissent avec des morphologies très allongées pour construire un réseau neuronal, tandis que les globules rouges sont petits et déformables pour pouvoir s’écouler dans nos vaisseaux sanguins. Les questions qui fascinent notre équipe sont de comprendre comment les cellules définissent leur forme particulière, et comment les informations de la géométrie cellulaire peuvent être véhiculées dans la fonction cellulaire ou dans l’organisation de l’intérieur de la cellule? Pour répondre à ces questions fondamentales, nous intégrons des méthodes originales à la frontière entre la biologie moléculaire, la génétique et la physique et les mathématiques. L’un de nos objectifs ultimes est de capturer le comportement biologique et la morphogénèse avec des modèles mathématiques. Notre recherche est articulée autour de deux axes principaux :
1- POSITIONNEMENT DU PLAN DE DIVISION.
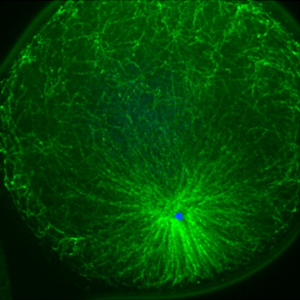
Le bon positionnement du site de division cellulaire est crucial non seulement pour la survie de toutes les cellules, mais aussi pour le développement de tissus multicellulaires. Notre laboratoire est fasciné par le processus d’embryogenèse précoce au cours duquel l’ovule est fécondé et suit une chorégraphie stéréotypée de divisions successives, appelées patrons de clivage. Notre objectif est de comprendre, prédire et établir des lois quantitatives qui expliquent les géométries des divisions cellulaires chez les embryons précoces. Pour cela, nous combinons des approches de modélisation mathématique, d’imagerie et de biophysique (Figure 1). Notamment, nous avons des méthodes permettant de contrôler la position et l’orientation du plan de division reposant sur l’utilisation de billes magnétiques permettant d’appliquer des forces sur la machinerie de division. Notre système expérimental modèle est l’embryon d’oursin (Vidéo 1), qui intègre de nombreux avantages pour l’étude du développement précoce. Pour étendre nos découvertes à d’autres contextes multicellulaires, nous collaborons avec d’autres laboratoires, travaillant sur des tissues mammifères ou des embryons d’autres espèces (Mouches, grenouilles…).
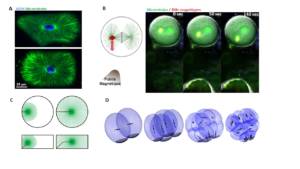
Figure 1: A. Chez le zygote d’oursins comme dans de nombreuses cellules, de larges astres de microtubules permettent de sentir la forme cellulaire et d’orienter le noyau et l’axe de division avec la forme de lacellule (Minc et al. 2011). B. Utilisation de pinces magnétiques pour appliquer des forces aux seins de cellules intactes et de contrôler la division cellulaire (Xie et al. 2021). C. Simulation numérique des forces exercées par les microtubules pour center le noyau en fonction de la forme cellulaire (Tanimoto et al. 2016). D. Simulation des géométries successives d’un embryon d’oursins (Pierre et al. 2016).
Légende : Embryons d’oursins se développant, dans lesquels l’ADN est marqué et suivi par fluorescence
2- FORME ET CROISSANCE CELLULAIRE.
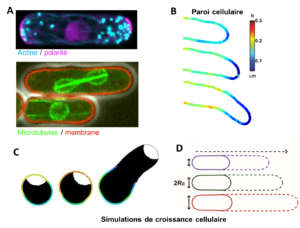
La morphologie des cellules individuelles ainsi que celles formant les tissus sous-tendent bon nombre de leurs fonctions. Nous cherchons à expliquer comment les cellules développent leur forme particulière et leur mode de croissance. Par exemple, de nombreuses cellules ne se développent qu’à un certain endroit, un processus appelé croissance polarisée, qui implique des réactions biochimiques complexes qui permettent de regrouper un domaine d’activité polarisé sous-cellulaire (Vidéo 2). Pour étudier ces aspects, nous utilisons des modèles de levures et de champignons, comme modèles de cellules très polarisées, qui grandissent rapidement et qui sont facilement manipulable génétiquement (Figure 2). Nous développons une recherche visant à comprendre comment les forces et les propriétés mécanique à la surface et au sein de la cellule interagissent avec des régulateurs biochimique pour définir morphogénèse et croissance.
Figure 2: A. Images de fluorescence de levures exprimant des marqueurs pour localiser et étudier le cytosquelette, la polarité la croissance et la division cellulaire (Bonazzi et al. 2015). B. Images super-résolues de l’épaisseur de la paroi cellulaire autour d’un hyphe grandissant de champignon de l’espèce aspergillus (Chevallier et al). C. Simulation numérique d’une spore de levure et de sa paroi (couleur) et polarité (blanc). D. Simulation de la vitesse de croissance.
Croissance et division de levures exprimant des marqueurs fluorescents d’enveloppe nucléaire et de synthèse de paroi.
Membres
 Maria Isabel ARJONA HIDALGO, Postdoctorante, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Maria Isabel ARJONA HIDALGO, Postdoctorante, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Camille BOUCHER, Ingénieure en biologie, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Camille BOUCHER, Ingénieure en biologie, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Serge DMITRIEFF, Chercheur, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Serge DMITRIEFF, Chercheur, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Amir KHOSRAVANIZADEH, Postdoctorant, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Amir KHOSRAVANIZADEH, Postdoctorant, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Charlotte MALLART, Postdoctorante, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Charlotte MALLART, Postdoctorante, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Elyn MANSOUR ROBIC, MINC LAB+33 (0)157278052, bureau 316B
Elyn MANSOUR ROBIC, MINC LAB+33 (0)157278052, bureau 316B Celia Maria MUNICIO DIAZ, Ingénieure en biologie, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Celia Maria MUNICIO DIAZ, Ingénieure en biologie, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Aude NOMMICK, Postdoctorante, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Aude NOMMICK, Postdoctorante, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Yannis REIGNIER, Doctorant, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Yannis REIGNIER, Doctorant, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Laura RUIZ, Ingénieure en biologie, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Laura RUIZ, Ingénieure en biologie, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Jérémy SALLE, Chercheur, MINC LAB+33 (0)1 57 27 80 52, bureau 316B
Jérémy SALLE, Chercheur, MINC LAB+33 (0)1 57 27 80 52, bureau 316B Jiawei XU, MINC LAB+33 (0)157278052, bureau 316B
Jiawei XU, MINC LAB+33 (0)157278052, bureau 316B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
- Xie J, Najafi J, Le Borgne R, Verbavatz J-M, Durieu C, Sallé J, and Minc N (2022) “Contribution of cytoplasm viscoelastic properties to mitotic spindle positioning” , Proc Natl Acad Sci U S A, 119 (8) e2115593119.
- Neeli-Venkata R, Municio Diaz C, Celador R, Sanchez Y, Minc N (2021) “Detection of surface forces by the cell-wall mechanosensor Wsc1 in yeast” , Developmental Cell 56, 1–15
- Palenzuela H, Lacroix B, Sallé J, Minami K, Shima T, Jegou A, Romet-Lemonne#G and Minc# N (2020) “In Vitro Reconstitution of Dynein Force Exertion in a Bulk Viscous Medium” , Current Biology, 30, 1–7
- Davi V, Chevalier L, Guo H, Tanimoto H, Barrett K, Couturier E, Boudaoud#A, and Minc# N, (2019) “Systematic mapping of cell wall mechanics in the regulation of cell morphogenesis” , Proc. Acad. Sci. USA, 116(28):13833-13838
- Sallé J, Xie J, Ershov D, Lacassin M, Dmitrieff S and Minc N, (2019) “Asymmetric division through a reduction of microtubule centering forces” J Cell Biol. 218(3):771-782 .
- Tanimoto H, Sallé J, Dodin L and Minc N (2018) “Physical forces determining the persistency and centring precision of microtubule asters” Nature Physics, 114, 848–854.
- Davì V, Tanimoto H, Ershov D, Haupt A, De Belly H, Le Borgne R, Couturier E, Boudaoud#A and Minc#, (2018) “Mechanosensation Dynamically Coordinates Polar Growth and Cell Wall Assembly to Promote Cell Survival” Developmental Cell, 45, 2, p170–182.
- Pierre A, Sallé J, Wühr M, Minc N., (2016) “Generic Theoretical Models to Predict Division Patterns of Cleaving Embryos.” Developmental Cell. 39(6):667-682
- Bonazzi* D, Julien* JD, Seddiki R, Romao M, Piel M, Boudaoud#A, Minc# N, (2014) “Symmetry breaking in spore germination relies on an interplay between polar cap stability and spore wall mechanics.” Developmental Cell, 28 (5):534-546
- Minc#, Burgess, D. and Chang F. (2011), “Influence of cell geometry on division plane positioning” Cell., 144 (3): 414-426.
Publications
Preprints
Revues
Chapitre de livre
Daria Bonazzi (2015)
Anaëlle Pierre (2017)
Valeria Davì (2018)
Héliciane Palenzuela (2020)
Jing Xie (2022)
Guillaume Romet-Lemonne (IJM) ; Delphine Delacour (IJM) ; Aki Kimura (NIG, Japan) ; Yohanns Bellaiche (Institut Curie, Paris) ; CP Heisenberg (IST austria) ; Martin Wühr (Princeton University) ; Dan Levy (U. Wyoming).
Arezki Boudaoud (Ecole Polytechnique); Etienne Couturier (MsC, Paris); Yolanda Sanchez (U. Cordoba); Miguel Peñalva (U. Madrid).
La Ligue Contre le Cancer ; Fondation Bettencourt-Schueller ; European Research Council ; Mairie De Paris; ANR ; Fondation de la Recherche Médicale ; FungiBrain (European ITN) FP7.
-
24/04/2024 : Mécanobiologie : la pression créatrice, contribution de l’équipe Minc à la publication d’un article dans le journal du CNRS