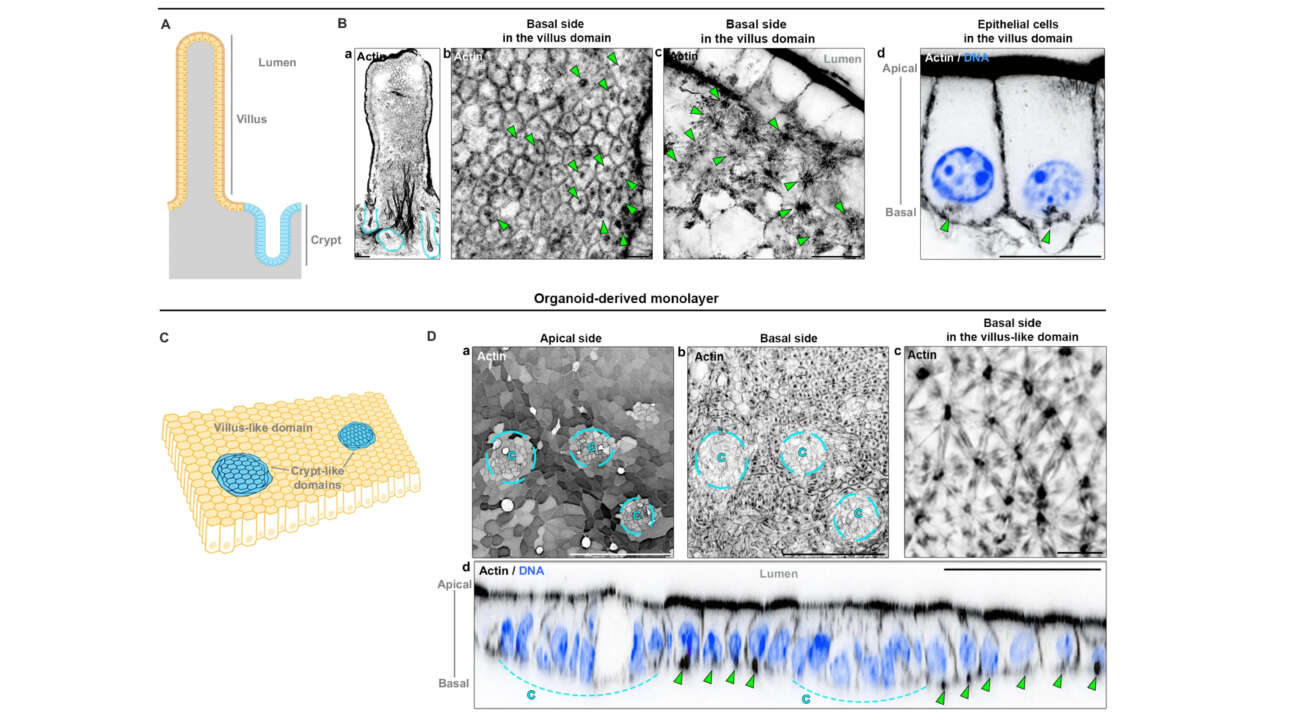
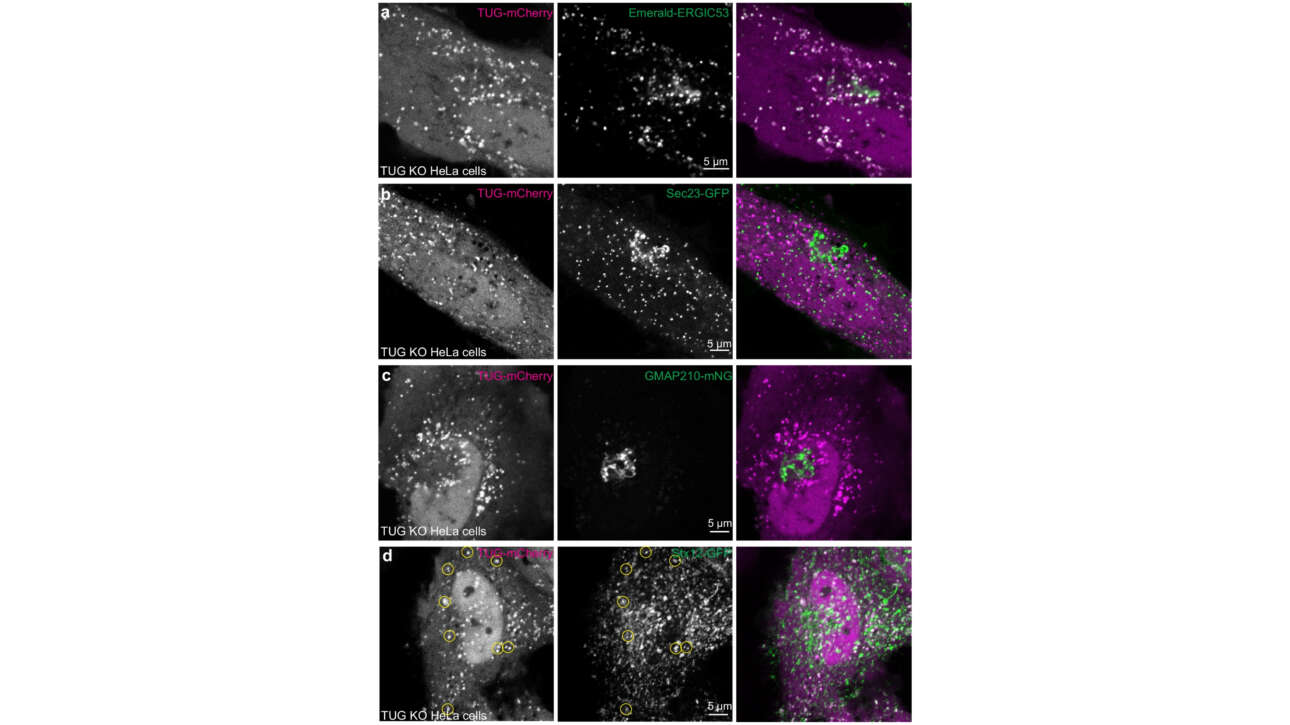
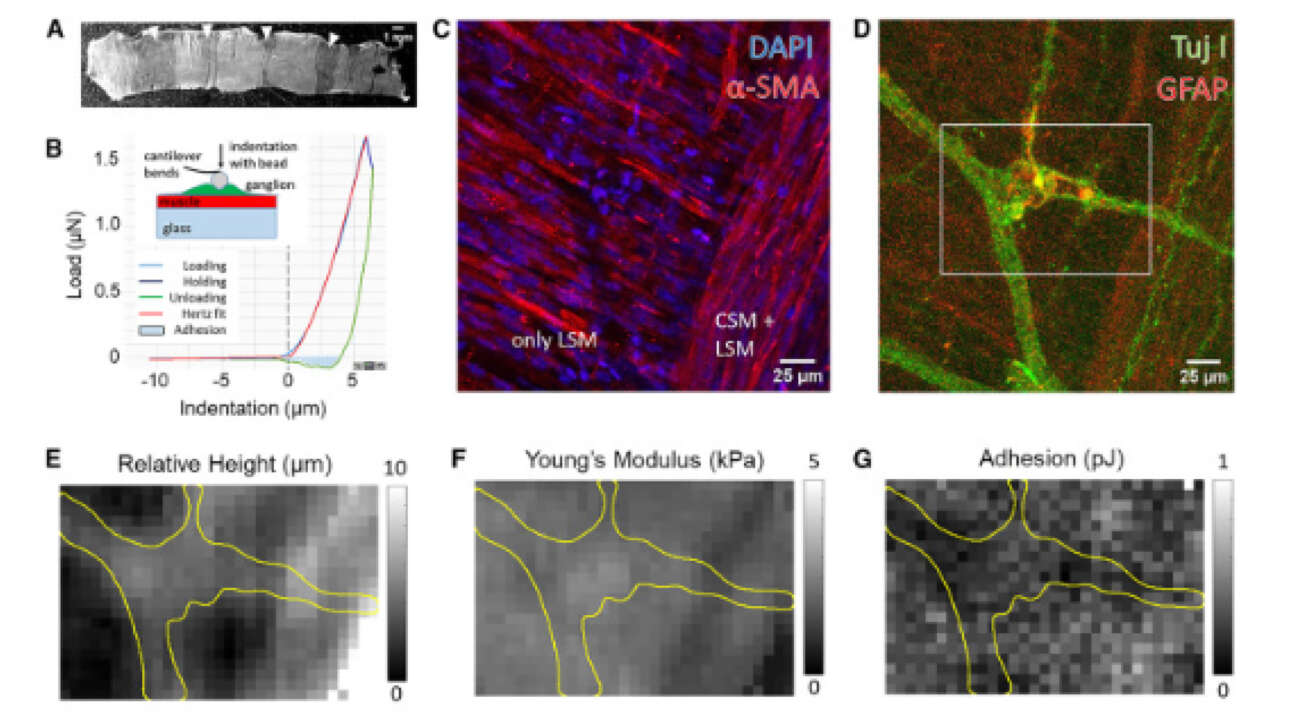
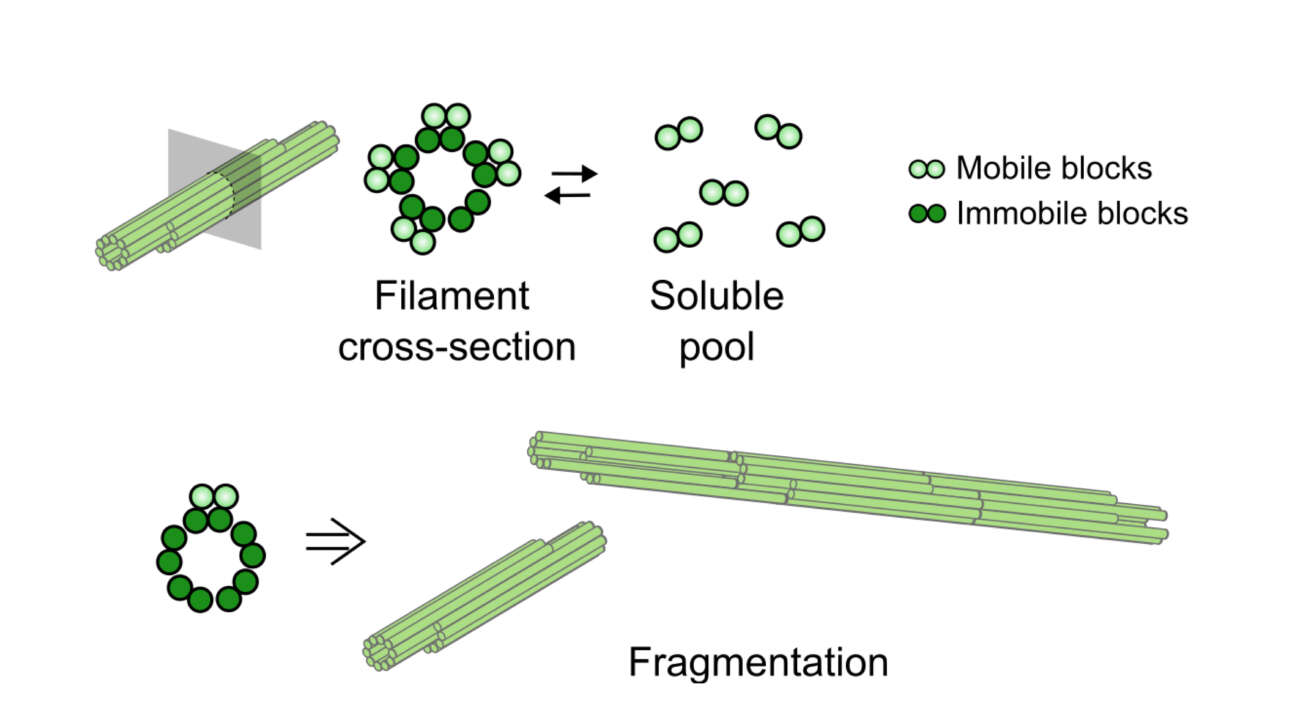
L'équipe Ladoux/Mege a contribué à la publication d'un nouvel article dans Nature Communications :
A multicellular star-shaped actin network underpins epithelial organization and connectivity
Résumé :
Epithelial tissues withstand external stresses while maintaining structural stability. Bicellular junctions and the actomyosin network support epithelial integrity, packing and remodelling. While their role in development and disease are well studied,…