Domaines chromatiniens et réplication
MARIE-NOËLLE PRIOLEAU
Au cours du développement, des domaines chromosomiques d’expression se structurent alors que les cellules se divisent activement. Avant chaque division, les cellules doivent copier leur génome le plus fidèlement possible sous peine de mort cellulaire ou d’instabilité génique pouvant conduire à l’établissement de cellules cancéreuses. Ce processus se nomme la réplication de l’ADN et démarre en de multiples sites spécifiques (~100 000 chez l’homme) ou à un site unique dans les bactéries, appelés origines de réplication.
L’équipe a deux axes de recherche : Initiation de la réplication chez les vertébrés (Marie-Noëlle Prioleau) et l’initiation de la réplication dans les bactéries (Jean-Luc Ferat).
Mots-clés : Réplication de l’ADN, initiation de la replication, G-quadruplex, Chromatine, sites Fragile, Organoïde Corticale, bacterie, co-helicase replicative
+33 (0)1 57 27 81 02 Contact @MariePrioleau
Initiation de la réplication chez les vertébrés (Marie-Noëlle Prioleau)
Dans les eucaryotes, un programme spatio-temporel contrôle le positionnement et le moment d’activation des origines de réplication au cours de la phase S du cycle cellulaire. Le but de l’équipe est d’identifier les mécanismes moléculaires qui mettent en place ce programme. Plus récemment nous cherchons également à comprendre comment deux mécanismes fondamentaux, la transcription et la réplication, agissent sur la même molécule simultanément de façon la plus harmonieuse possible. En effet, il est maintenant clairement établi que des conflits entre ces deux machineries peuvent être source d’instabilité génique.
Afin d’avoir une vision générale de la réplication du génome humain, nous développons des analyses à haut débit visant à cartographier les origines de réplication ainsi que leur moment d’activation (Figure 1).
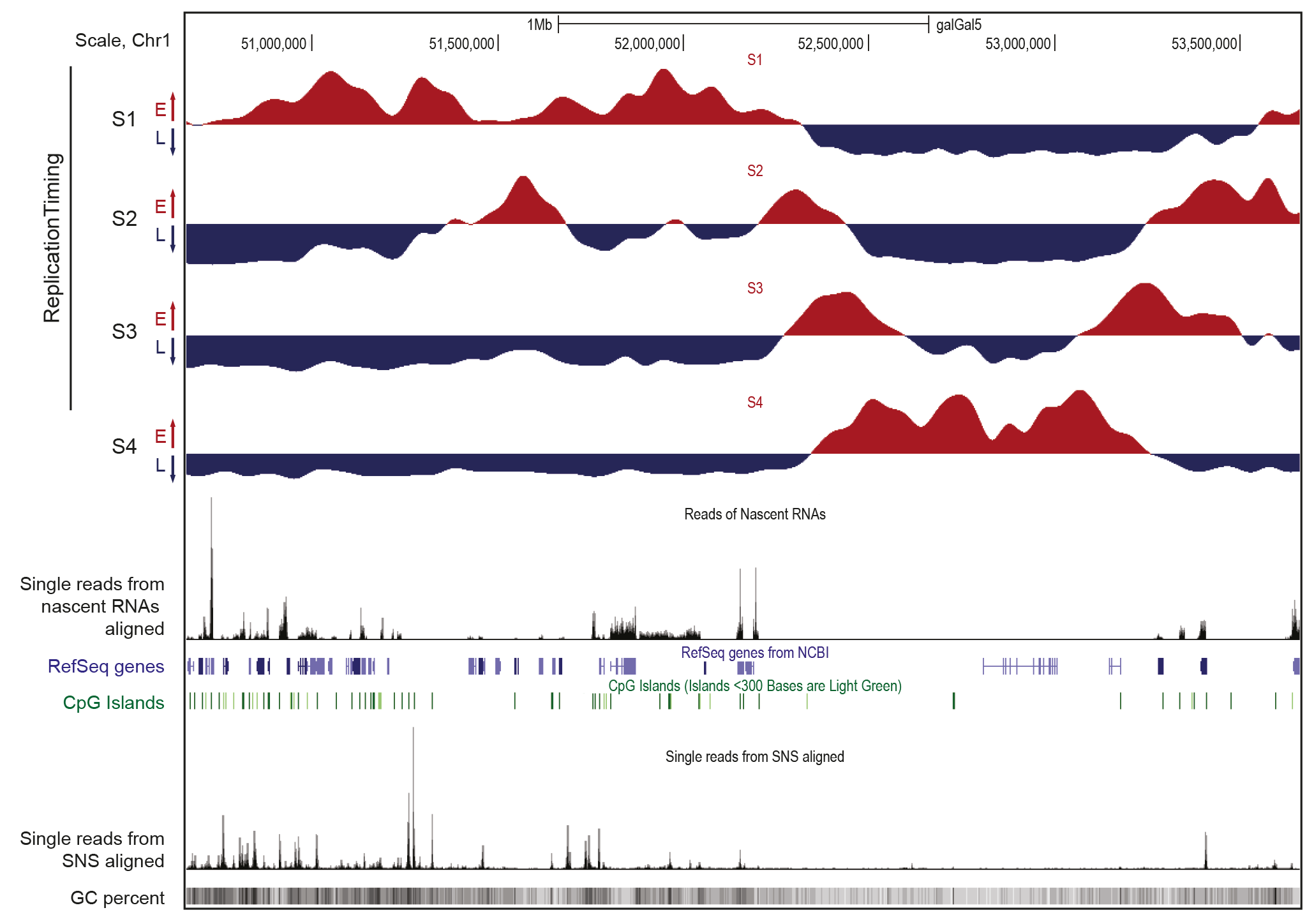
Figure 1. Caractéristiques des domaines de réplication précoce et tardive
Visualisation sur le navigateur UCSC d’une région génomique de 3Mb du chromosome 1 du poulet. Les données ont été obtenues dans la lignée cellulaire aviaire DT40. Les profils d’enrichissement en brins naissants (NS) dans les quatre fractions de la phase S, de précoce à tardive, sont présentés séparément (S1 à S4). Les régions enrichies en NS et appauvries pour chaque fraction sont indiquées en rouge et en bleu, respectivement. Ces données indiquent le programme temporel de réplication. Les profils d’ARN naissants et de SNS (petits brins naissants marquant les origines de réplication, programme spatial de réplication) purifiés à partir de cellules asynchrones sont montrés. Entre ces données sont indiqués les gènes annotés (gènes RefSeq) et les îlots CpG. Enfin, le contenu en GC (pourcentage GC) est indiqué en bas de la figure.
Une collaboration avec des statisticiens et bio-informaticiens nous a permis d’établir des liens entre ces cartes et des données génomiques sur la structure de la chromatine et l’expression des gènes. Nous utilisons également un modèle cellulaire aviaire (la lignée DT40) qui a la propriété unique d’effectuer très efficacement la recombinaison homologue. Ce modèle génétique puissant nous permet de tester très efficacement différentes hypothèses extraites des analyses génomiques. Nous avons pu ainsi montrer que les G-quadruplex jouaient un rôle clé dans le déclenchement des origines de réplication. Nous venons d’identifier un motif complexe que l’on retrouve dans la moitié des origines fortes chez l’humain, la souris et le poulet. Les projets futurs s’attacheront à comprendre comment ce motif assure le recrutement efficace de la machinerie de réplication.
Dans un deuxième axe de recherche, nous analysons comment le signal origine porté par ce motif complexe interagit avec le processus de transcription qui s’opère sur le même substrat, l’ADN. Ces analyses nous permettront de tester une hypothèse largement étayée selon laquelle la transcription est capable de déplacer les points de départ de la réplication et ainsi de favoriser de manière générale l’initiation de la réplication dans les régions intergéniques. Les sites fragiles communs (SFC) sont des sites récurrents de réarrangements chromosomiques dans les cancers et certaines pathologies neurologiques. Ils se trouvent au sein de grands gènes (> 300 kb) transcrits et répliqués en fin de phase S. Une hypothèse concernant leur formation est que la pauvreté en événements d’initiation de la réplication, le long de ces gènes, entrainerait une réplication incomplète de ces régions avant la mitose. La transcription enlèverait les complexes de pré-réplication fixés dans le corps de ces gènes induisant ainsi une déplétion en origines de réplication. Nous cherchons à tester la capacité de l’ARN polymérase II à déplacer les complexes de pré-réplication en insérant un promoteur fort ou inductible en amont d’une origine modèle minimale très efficace dépourvue d’activité transcriptionnelle. Ce projet permettra d’explorer une hypothèse importante sur la formation des SFC. Sur le plus long terme, nous testerons l’hypothèse selon laquelle les sites récurrents de cassures observés durant la prolifération de précurseurs neuronaux ont les propriétés caractéristiques des SFC. Pour cela nous utiliserons comme système modèle des organoïdes cérébraux humains.
La compréhension du mode de duplication des génomes eucaryotes est essentielle. En effet, la réplication assure non seulement le maintien de l’intégrité des génomes, mais également coordonne la mise en place de programmes d’expression au cours du développement.
L’initiation de la réplication dans les bactéries (Jean-Luc Ferat)
Dans les bactéries, la réplication est initiée à une origine unique par la protéine initiatrice, DnaA. La liaison ordonnée de DnaA à l’origine conduit à l’ouverture local de la double helice d’ADN et au recrutement des hélicases réplicatives directement sur l’ADN simple brin. Cette étape est fortement régulée et assistée par une co-hélicase, qui synchronise le chargement des hélicases réplicatives, assurant ainsi l’initiation bidirectionnelle de la réplication (BRI). Nous cherchons à comprendre 1) comment la co-hélicase DciA active de manière synchrone les hélicases réplicatives pendant l’initiation de la réplication et 2) comment la réplication initiée en absence de DciA conduit à la dégradation pathologique du chromosome entier. Nous avons développé un large éventail d’approches expérimentales (cytologiques, génétique, génomique, bioinformatique) pour répondre à ces deux questions. Nous utilisons Escherichia coli et Vibrio cholerae comme organismes modèles.
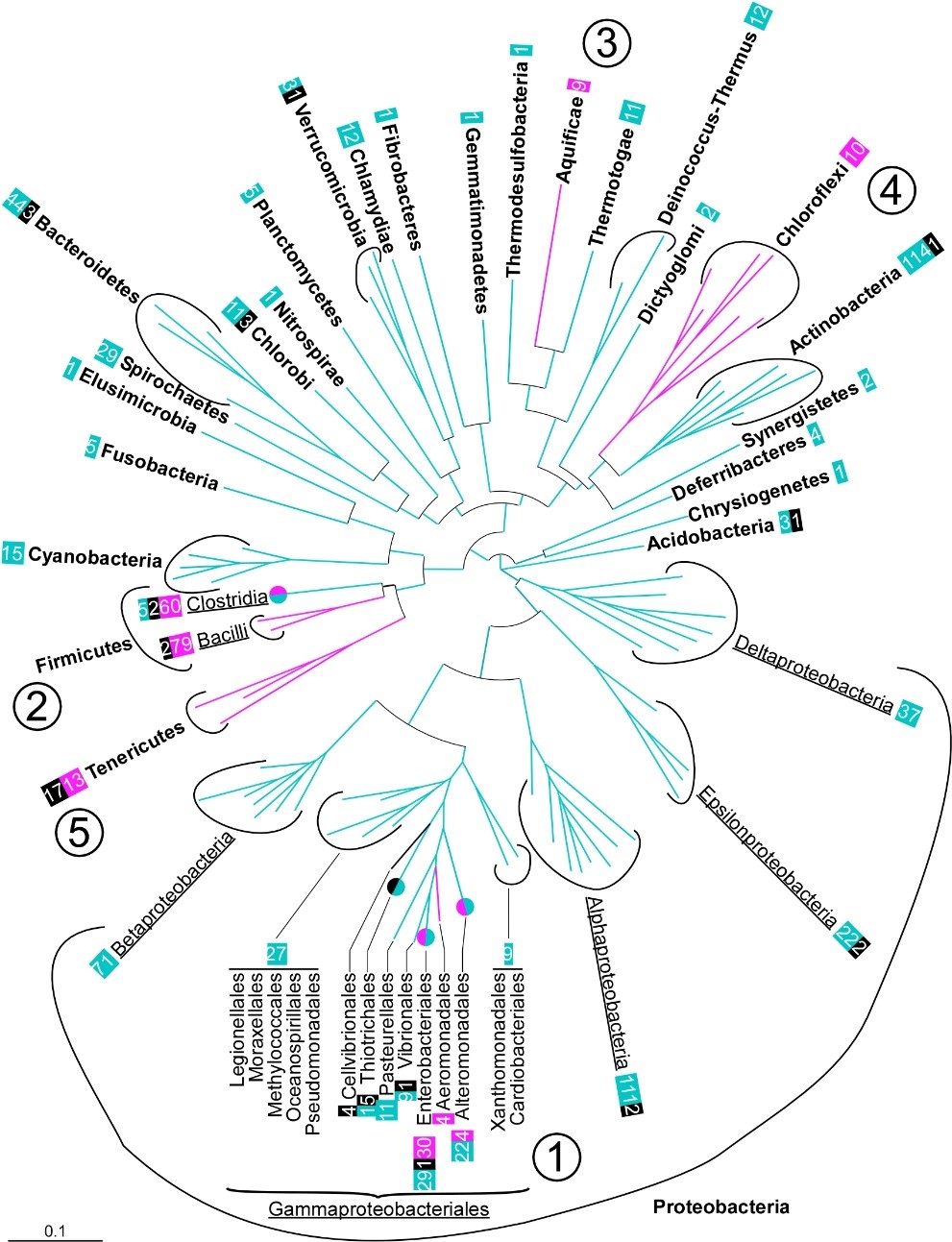
Fig.1 Distribution of domesticated dnaC and dciA in the bacterial domain. Distribution of the bacterial phyla investigated on a 16S rRNA-based circular phylogenetic tree (Methods). When the presence of dnaC/I or dciA within a phylum (bold) was not homogeneous, classes (underlined) or orders (normal) are specified. Numbers boxed refer to the number of species’ genomes (and not strains) containing domesticated dnaC/I (fuchsia), dciA (turquoise) or neither (black). fuchsia / turquoise circles: orders in which dciA and dnaC/I genes were identified. The order Thiotrichales is identified with a green/black circle because most species lack dciA and dnaC/I. ① Gammaproteobacteria, ② Firmicutes, ③ Aquificae, ④ Chloroflexi and ⑤ Tenericutes.
In bacteria, replication is initiated at a single origin by the initiator protein, DnaA. The ordered binding of DnaA at the origin creates a local unwinding of the double helix that enables the recruitment of the replicative helicase, DnaB. Replisomes are then assembled on this nucleoprotein platform to trigger Bidirectional Replication Initiation. Contrary to eukaryotes, bacteria load directly their helicase on single strand DNA during replication initiation. This process is highly regulated and assisted by co-helicases in the three domains of life. Bacterial co-helicases are diverse. The first family that was studied, DnaC/I (DnaC in Escherichia coli and DnaI in Bacillus subtilis), is present in only a few orders of bacteria and results from the domestication of a phage gene (Brézellec et al., 2016). The main family of co-helicase is DciA. It was discovered only a few years ago although it is present in the vas majority of bacterial orders (Figure 1). We established that DciA, is the ancestral bacterial co-helicase (Brézellec et al., 2016, 2017).
We conducted a biochemical investigation of DciA together with its cognate replicative helicase to compare its activity to that of DnaC of E. coli. This study revealed surprisingly that contrary to DnaC/I the loading of the replicative helicase is not dependent on DciA. In other words, the replicative helicase of dciA-sepcies can self-load on DNA. At most, DciA improves the loading efficiency (Figure 2) (Marsin et al., 2021).
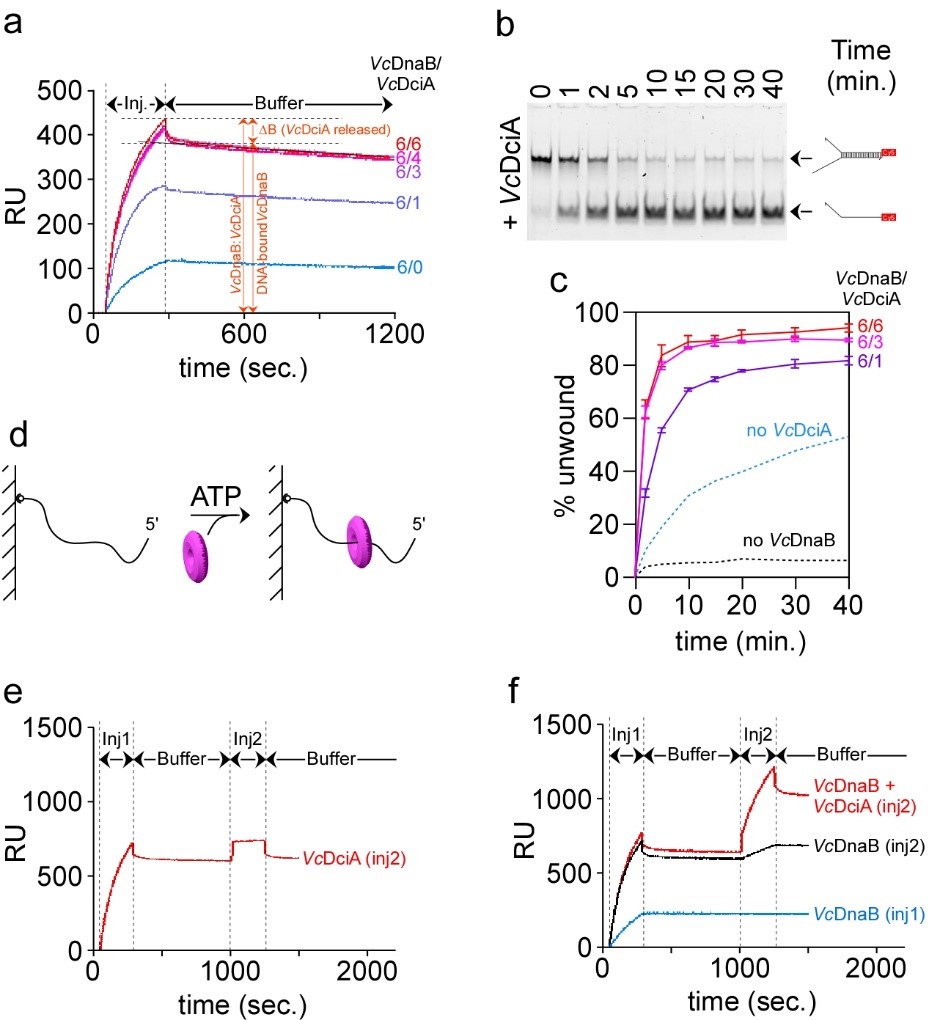
Fig.2 VcDciA stimulates the loading of VcDnaB on DNA. a, SPR analysis of the loading of VcDnaB on DNA in presence of VcDciA. Experiments were conducted as described in Figure 1B in presence of VcDnaB at 625nM. Different VcDnaB/VcDciA ratios (from 6/0 to 6/6 in subunits) were analyzed. ΔB represents the mass of VcDciA proteins that dissociated immediately after the loading of the VcDciA•VcDnaB complex on DNA. b, DNA unwinding kinetics in presence of VcDciA. The experiments were carried out as described in Figure 1D, except that VcDciA (variable concentrations) was added to VcDnaB (625 nM). The types of migration products are indicated on the right. c, DNA unwinding quantification in presence of VcDciA. The procedure is the same as that described in Figure 1E. The error bars correspond to the mean +/- SEM of three independent experiments. d, Helicase loading in absence of Mg2+. The experiment is carried out in absence of Mg to prevent the activation of the helicase and its translocation on DNA in the 5’ → 3’ direction until its release. ATP was added for it is required for the hexamerization of the helicase.
Flow cytometry analysis of [dciA–] cells confirmed that the replicative helicase of V. cholerae can load itself in vivo without DciA. A genomic approach intended to assessing the replication status of [dciA–] cells revealed that DciA is essential to ensure Bidirectional Replication Initiation (BRI), likely by synchronizing the loading of the replicative helicase during replication initiation. We showed that in absence of DciA, replication is initiated unidirectionally and that this Unidirectional Replication Initiation (URI) is infallibly followed by a catastrophic sequence of events ending in chromosome degradation (Besombes et al. submitted) (Figure 3).
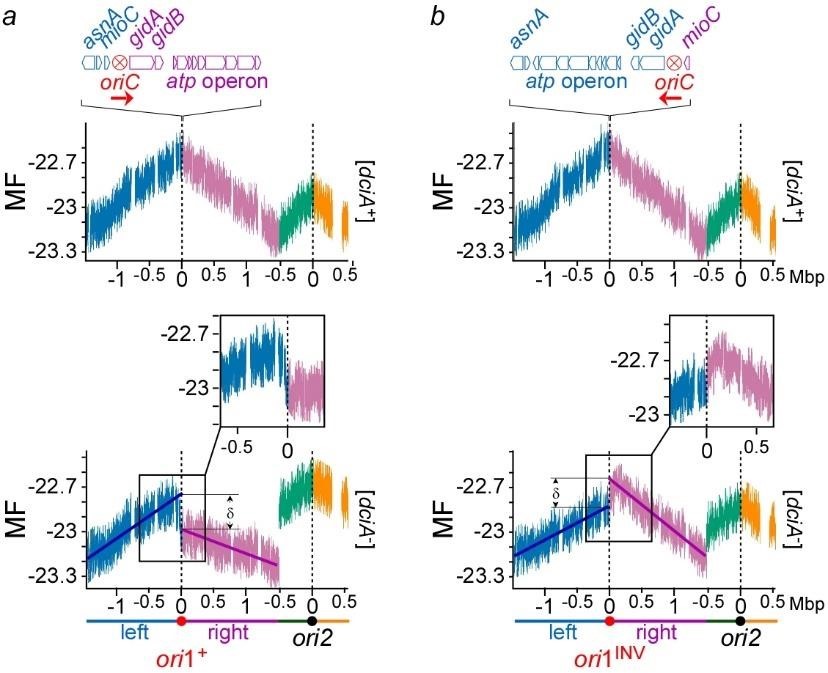
Fig.3 Replication is initiated unidirectionally at ori1 on Chr1 in [dciA–] cells. a) MF profiles of ori1+ dciA+ (top )and ori1+ [dciA–] populations (bottom) maintained in continuous growth in minimal medium at 30°C for 12h before DNA extraction and sequence. The reads were aligned against V. cholerae reference genome (using Burrows-Wheeler Aligner) to assign a number of reads per genome position (marker frequency). The value for each position of the genome corresponds to the marker frequency average of 1000 consecutive positions (1kb) surrounding it. log2 of these values are used to correct for the exponential growth of the cells (MF). b) same as in a), except that the origin of replication was inverted (oriINV). The left and right replichore of Chr1 and Chr2 are colored in blue, purple, green and orange, respectively.
Our research program is aimed now at understanding how unprogrammed URI leads to chromosome degradation and how DciA loads and activates synchronously the replicative helicases during replication initiation. We address these questions 1) by inventorying the diverse routes followed by [dciA–] cells and dissecting them frame by frame through a large diversity of approaches (fluorescent microscopy, digital PCR, PFGE, NGS, etc.), 2) by deciphering the functional network of DciA (two hybrid, Tn-seq) and 3) by creating a strain of V. cholerae in which dciA is replaced by dnaC to trace the modifications and their succession during adaptation to a new co-helicase and eventually to understand the mechanism underlying BRI.
We use two bacterial model organisms (Vibrio cholerae and Escherichia coli) to address these two questions, and developed a large spectrum of experimental approaches (cytological, genetic, genomic, bioinformatic).
References
Besombes A, Adam Y, Possoz C, Junier Y, Barre FX, Ferat JL. (submitted) Bidirectional replication initiation prevents chromosome degradation
Marsin S, Adam Y, Cargemel C, Andreani J, Baconnais S, Legrand P, Li de la Sierra-Gallay I, Humbert A, Aumont-Nicaise M, Velours C, Ochsenbein F, Durand D, Le Cam E, Walbott H, Possoz C, Quevillon-Cheruel S, Ferat JL. (2021) Study of the DnaB:DciA interplay reveals insights into the primary mode of loading of the bacterial replicative helicase. Nucleic Acids Res. 49:6569-6586
Brézellec P, Petit MA, Pasek S, Vallet-Gely I, Possoz C, Ferat JL. (2017) Domestication of Lambda Phage Genes into a Putative Third Type of Replicative Helicase Matchmaker. Genome Biol Evol. 9:1561-1566
Brézellec P, Vallet-Gely I, Possoz C, Quevillon-Cheruel S, Ferat JL. (2016) DciA is an ancestral replicative helicase operator essential for bacterial replication initiation. Nat Commun. 7:13271[
Membres
 Theo BARET, Biology engineer, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Theo BARET, Biology engineer, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B Amélie BESOMBES, PhD student, PRIOLEAU LABbureau 520B
Amélie BESOMBES, PhD student, PRIOLEAU LABbureau 520B Caroline DONCARLI, Biology engineer, PRIOLEAU LAB+33 (0)1 57 27 81 24, bureau 520B
Caroline DONCARLI, Biology engineer, PRIOLEAU LAB+33 (0)1 57 27 81 24, bureau 520B Kathrin MARHEINEKE, Researcher, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Kathrin MARHEINEKE, Researcher, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B Aurélie MASSON, PhD student, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Aurélie MASSON, PhD student, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
1) Clustering of strong replicators associated with active promoters is sufficient to establish an early-replicating domain. Brossas C, Valton AL, Venev SV, Chilaka S, Counillon A, Laurent M, Goncalves C, Duriez B, Picard F, Dekker J, Prioleau MN. EMBO J. 2020 Nov 2;39(21):e99520. doi: 10.15252/embj.201899520. Epub 2020 Sep 16. PMID: 32935369
2) Replication dynamics of individual loci in single living cells reveal changes in the degree of replication stochasticity through S phase. Duriez B, Chilaka S, Bercher JF, Hercul E, Prioleau MN. Nucleic Acids Res. 2019 Jun 4;47(10):5155-5169. doi: 10.1093/nar/gkz220. PMID: 30926993
3) Evolution of replication origins in vertebrate genomes: rapid turnover despite selective constraints. Massip F, Laurent M, Brossas C, Fernández-Justel JM, Gómez M, Prioleau MN, Duret L, Picard F. Nucleic Acids Res. 2019 Jun 4;47(10):5114-5125. doi: 10.1093/nar/gkz182. PMID: 30916335
4) Transcription-dependent regulation of replication dynamics modulates genome stability. Blin M, Le Tallec B, Nähse V, Schmidt M, Brossas C, Millot GA, Prioleau MN, Debatisse M. Nat Struct Mol Biol. 2019 Jan;26(1):58-66. doi: 10.1038/s41594-018-0170-1. Epub 2018 Dec 31. PMID: 30598553
5) The spatiotemporal program of DNA replication is associated with specific combinations of chromatin marks in human cells. Picard F, Cadoret JC, Audit B, Arneodo A, Alberti A, Battail C, Duret L, Prioleau MN. PLoS Genet. 2014 May 1;10(5):e1004282. doi: 10.1371/journal.pgen.1004282. eCollection 2014 May. PMID: 24785686
6) G4 motifs affect origin positioning and efficiency in two vertebrate replicators. Valton AL, Hassan-Zadeh V, Lema I, Boggetto N, Alberti P, Saintomé C, Riou JF, Prioleau MN. EMBO J. 2014 Apr 1;33(7):732-46. doi: 10.1002/embj.201387506. Epub 2014 Feb 12. PMID: 24521668
7) USF binding sequences from the HS4 insulator element impose early replication timing on a vertebrate replicator. Hassan-Zadeh V, Chilaka S, Cadoret JC, Ma MK, Boggetto N, West AG, Prioleau MN. PLoS Biol. 2012;10(3):e1001277. doi: 10.1371/journal.pbio.1001277. Epub 2012 Mar 6. PMID: 22412349
8) Genome-wide studies highlight indirect links between human replication origins and gene regulation. Cadoret JC, Meisch F, Hassan-Zadeh V, Luyten I, Guillet C, Duret L, Quesneville H, Prioleau MN. Proc Natl Acad Sci U S A. 2008 Oct 14;105(41):15837-42. doi: 10.1073/pnas.0805208105. Epub 2008 Oct 6. PMID: 18838675
9) Broadening of DNA replication origin usage during metazoan cell differentiation. Dazy S, Gandrillon O, Hyrien O, Prioleau MN. EMBO Rep. 2006 Aug;7(8):806-11. doi: 10.1038/sj.embor.7400736. Epub 2006 Jun 16. PMID: 16799461
10) Replication of the chicken beta-globin locus: early-firing origins at the 5′ HS4 insulator and the rho- and betaA-globin genes show opposite epigenetic modifications. Prioleau MN, Gendron MC, Hyrien O. Mol Cell Biol. 2003 May;23(10):3536-49. doi: 10.1128/MCB.23.10.3536-3549.2003. PMID: 12724412
Publications
Revues
- « Rôle de l’insulateur 5’HS4 du poulet dans la régulation de la réplication » soutenue en Juin 2009 par Vahideh Hassan-Zadeh.
- « Mécanismes moléculaires de l’initiation de la réplication » soutenue en Décembre 2010 par Françoise Meisch.
- « Identification de séquences cis-nucléiques nécessaires à l’initiation de la réplication chez les vertébrés » soutenue en Juin 2014 par Anne-Laure Valton.
- « Construction d’un domaine synthétique de réplication précoce et impact sur la structure chromatinienne et la permissivité transcriptionnelle » soutenue en Septembre 2015 par Caroline Tonnerre-Doncarli Brossas.
- « Mécanismes moléculaires impliqués dans la régulation du moment de déclenchement des origines de réplication » soutenue en Novembre 2016 par Antonin Counillon.
- « Rôle des G-quadruplexes dans la spécification des origines de réplication chez les vertébrés » soutenue en Septembre 2016 par Marc Laurent.
- « Etude moléculaire des éléments cis-régulateurs et de l’organisation de la chromatine des origines de réplication » soutenue en Septembre 2020 par Jérémy Poulet-Benedetti.
Nationales :
- Imagerie cellulaire et cytométrie en flux : Plateforme Imago-Seine, Institut Jacques Monod
- Organoïdes corticaux : Plateforme enSCORE, Labex Who am I ?
- Bio-informatique et bio-statistique : LBBE, UCB Lyon 1 (Laurent Duret) et ENS de Lyon (Franck Picard)
- G-quadruplex : Inserm U565, CNRS UMR 7196, MNHN, Paris, France (Patrizia Alberti, Carole Saintomé et Jean-François Riou)
- Sites Fragiles Communs : : Institut Gustave Roussy, Villejuif (Michelle Debatisse et Stéphane Koundrioukoff)
Internationales :
- Organisation tri-dimensionnelle du noyau : University of Massachusetts Medical School, Worcester, Etats-Unis (Anne-Laure Valton et Job Dekker)
- Rôle des G4s dans le positionnement des nucléosomes : Fox Chase Cancer center, Philadelphie, Etats-Unis et Moscow State University, Moscow, Russie (Vasily Studitsky).
Equipe-partenaire du Labex “Who am I?”


