Chromatin Dynamics in Mammalian Development
MAXIM GREENBERG
La méthylation de l’ADN est tune marque épigénétique stable absolument essentielle pour le développement des mammifères. Dans le laboratoire Greenberg, nous nous concentrons principalement sur la compréhension des mécanismes variés et des fonctions de la méthylation de l’ADN dans le contexte du développement. Nous prenons une approche multidisciplinaire utilisant la génétique la génomique et la biochimie. Nous aspirons à comprendre comment la méthylation de l’ADN interagit avec les autres voies de la chromatine pour obtenir une image holistique de la régulation génique médiée par la chromatine.
Mots clés : Méthylation de l’ADN, épigénétique, chromatine, cellules souches embryonnaires murines, développement
Office: +33 (0) 1 57 27 81 20 Lab: 80 32 Contact @maxvcg.bsky.social https://www.maximgreenberglab.com/
Objectif de recherche
Nous aspirons à obtenir une compréhension claire des conséquences épigénétiques profondes de la méthylation de l’ADN durant une fenêtre de développement ayant lieu durant la première semaine de l’embryogénèse chez la souris et la seconde chez l’humain, avec des répercussions tout au long de la vie.
Contexte
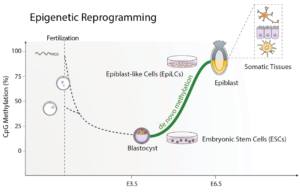
Immédiatement après la fertilisation, les génomes des mammifères subissent un réarrangement spectaculaire de l’épigénome, quand l’embryon effectue une transition du zygote vers les cellules pluripotentes s’apprêtant à s’engager dans un lignage. C’est le meilleur exemple de reprogrammation de la méthylation de l’ADN, puisque le modèle gamétique est totalement effacé, et l’embryon démarre une vague de méthylation de l’ADN de novo. De plus, une fois le modèle de méthylation de l’ADN établi, des mécanismes assurent un maintien fidèle de la marque durant la division cellulaire. Ainsi, la méthylation de l’ADN déposée dans l’embryon précoce a potentiellement un effet tout au long de la vie.
La méthylation de l’ADN est une modification typiquement associée à la répression génique et aux éléments répétés, et à une minorité de gènes codant pour des protéines. Nous avons précédemment décrit la régulation du gène Zdbf2 chez la souris, qui est programmée durant le programme de méthylation de l’ADN de novo. Remettant en question le paradigme, dans le cas la méthylation de l’ADN est requise pour l’activation d’un gène en antagonisant les protéines de mise sous silence du groupe polycomb. Si la méthylation de l’ADN échoue, le gène reste silencieux toute la vie et cela conduit à un phénotype de ralentissement de croissance.
Objectifs
Nous allons tenter de répondre à une question large : que fait la méthylation de l’ADN quand elle n’est pas aux promoteurs ? Et c’est corollaire : comment les promoteurs restent dépourvus de méthylation de l’ADN ? Il est connu depuis longtemps que la méthylation de l’ADN aux promoteurs denses en CpGs, aussi appelés promoteurs à ilots CpG (CGI), est associée à un état silencieux du gène. Cependant, le génome des mammifères est hautement méthylé avec pour exception des promoteurs CGI. Ce qui veut dire que la méthylation de l’ADN est importante pour éteindre seulement une petite proportion des gènes codant pour des protéines. La fonction de la méthylation de l’ADN aux régions par exemple intergéniques, ou à l’intérieur des gènes transcrits, est moins claire.

Sur la base de nous études sur Zdbf2, nous nous intéressons à la connexion clé qu’est l’antagonisme observé entre la méthylation de l’ADN et la machinerie de mise en silence polycomb. Nous sommes très motivés pour acquérir des connaissances sur les fondements mécanistes de la régulation génique basée sur la méthylation de l’ADN. De manière cruciale, nous voulons comprendre la relevance des formes de méthylation de l’ADN non canoniques sur le développement.
Méthodes
Dans le laboratoire Greenberg, les cellules souches embryonnaires murines (ESCs) sont notre terrain de jeu. Les ESCs offrent de nombreux avantages pour l’étude de la méthylation de l’ADN : elles sont résiliantes aux mutations dans les gènes des ADN méthyltransférases, elles sont dociles à l’édition du génome par CRISPR/Cas9 et aux cibles génétiques, et peuvent être facilement collectées en grande quantité pour des expériences robustes. De manière importante, nous utilisons un système de différentiation qui récapitule la reprogrammation de la méthylation de l’ADN qui a lieu in vivo. Comme nous avons fait pour Zdbf2, les découvertes que nous faisons dans notre système cellulaire seront complémentées par les modèles murins pour valider l’importance développementale. Nous nous baserons sur des approches de séquençage de nouvelle génération pour approfondir notre vision de la régulation de la méthylation de l’ADN. C’est un moment excitant dans le domaine de l’épigénétique, avec l’émergence d’outils d’édition de l’épigénome. En utilisant un ciblage basé sur le CRISPR/Cas9, nous pouvons explorer avec précision la fonction de la méthylation de l’ADN locus par locus.

Dans le futur, nous sommes excités d’employer des cribles génétiques basés sur le CRISPR/Cas9 pour découvrir de nouveaux facteurs, ainsi que de travailler avec la plateforme de protéomique haut de gamme de l’IJM pour des approches basées sur la spectrométrie de masse. Nous sommes toujours ouverts aux nouvelles technologies et idées, donc nos méthodes vont constamment évoluer ! Si vous êtes intéressés par notre recherche et souhaitez en savoir plus, n’hésitez pas à nous contacter !
Membres
 Angélique DAVID, PhD student, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B
Angélique DAVID, PhD student, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B Priscillia LHOUMAUD, Researcher, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B
Priscillia LHOUMAUD, Researcher, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B Marlet MORALES-FRANCO, Postdoctoral researcher, GREENBERG LAB
Marlet MORALES-FRANCO, Postdoctoral researcher, GREENBERG LAB Julie PAULIAT, Intern, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B
Julie PAULIAT, Intern, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B Margherita SCARPA, Biology engineer, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B
Margherita SCARPA, Biology engineer, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B Teresa URLI, PhD student, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B
Teresa URLI, PhD student, GREENBERG LAB+33 (0)1 57 27 81 20, bureau 542B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
Greenberg M.V.C. (2021) Get Out and Stay Out: New Insights into DNA Methylation Reprogramming in Mammals. Front. Cell Dev. Biol. 8:629068
Greenberg M.V.C, Bourc’his D. (2019) The diverse roles of DNA methylation in mammalian development and disease. Nature Reviews Molecular Cell Biology 20:590-607
Greenberg M.V.C.†, Teissandier A., Walter, M., Noordermeer D., Bourc’his D†. (2019) Dynamic enhancer partitioning instructs activation of a growth-related gene during exit from naïve pluripotency. eLife 8:e44057
Greenberg M.V.C.*, Glaser J.*, Borsos M., El Marjou F., Walter M., Teissandier A., Bourc’his D. (2017) Transient transcription in the early embryo sets an epigenetic state that programs postnatal growth. Nature Genet. 49:110-118
F1000Prime recommendation – F1000Prime.com/726971069#eval793526035
Greenberg M.V.C., Bourc’his D. (2015) Cultural relativism: maintenance of genomic imprints in pluripotent stem cell culture systems. Curr. Op. Genet. & Dev. 31:42–49
Greenberg M.V.C.*, Deleris A.*, Hale C.J.*, Liu A., Feng S., Jacobsen S.E. (2013) Interplay between active chromatin marks and RNA-directed DNA methylation in Arabidopsis thaliana. PLoS Genet. 9(11): e1003946, 1-11
Stroud H., Greenberg M.V.C., Feng S., Bernatavichute Y.V., Jacobsen S.E. (2013) Comprehensive analysis of silencing mutants reveals complex regulation of the Arabidopsis methylome. Cell 152: 352-64
Ausin I.*, Greenberg M.V.C.*, Simanshu D.K.*, Hale C.J., Vashisht A.A., Simon S.A., Lee T., Feng S., Espanola S.D., Meyers B.C., Wohlschlegel J.A., Patel D.J., Jacobsen S.E. (2012) INVOLVED IN DE NOVO 2 containing complex involved in RNA-directed DNA methylation in Arabidopsis. Proc. Nat. Acad. Sci. U. S. A. 109: 8374-8381
Greenberg M.V.C., Ausin I., Chan S.W.L., Cokus S.J., Cuperus J.T., Feng S., Law J.A., Chu C., Pellegrini M., Carrington J.C., Jacobsen S.E. (2011) Identification of genes required for de novo DNA methylation in Arabidopsis. Epigenetics6:344-354
Deleris A.*, Greenberg M.V.C.*, Ausin I., Law R.W.Y., Moissiard G., Schubert D., Jacobsen S.E. (2010) Involvement of a Jumonji-C domain-containing histone demethylase in DRM2-mediated maintenance of DNA Methylation. EMBO Rep.11: 950-955
Publication
Preprint
Revue
- ANR “REMEDY”:
- Pierre-Antoine Defossez (Coordinator), Epigenetics and Cell Fate, CNRS, Université Paris Cité, Paris
- Salvatore Spicuglia (Partner), TAGC, INSERM, Aix-Marseille University, Marseille
- LabEx Who Am I? “DECODERS”:
- Benoit Palancade (Coordinator), Université Paris Cité, Institut Jacques Monod, CNRS, Paris

Article CNRS INSB : Les signaux épigénétiques : comment les marques de l’ADN façonnent le plan de la vie
Le prix Georges Brahms de la Fondation CNRS attribué à Priscillia Lhoumaud


