Domaines chromatiniens et réplication
MARIE-NOËLLE PRIOLEAU
Au cours du développement, les cellules se divisent activement et organisent progressivement des domaines chromosomiques d’expression. Avant chaque division, elles doivent copier leur génome avec une très grande fidélité. Une erreur dans ce processus peut entraîner la mort cellulaire ou une instabilité génétique susceptible de conduire à l’apparition de cellules cancéreuses. Cette étape cruciale s’appelle la réplication de l’ADN. Elle débute en de nombreux sites spécifiques du génome — environ 100 000 chez l’humain — appelés origines de réplication.
Mots-clés : Réplication de l’ADN, initiation de la réplication, G-quadruplex, Chromatine, sites Fragile, Organoïde Cortical
+33 (0)1 57 27 81 02 Contact @MariePrioleau
Initiation de la réplication chez les vertébrés
Chez les eucaryotes, l’activation des origines de réplication est finement régulée dans l’espace et dans le temps au cours de la phase S du cycle cellulaire. Notre équipe cherche à identifier les mécanismes moléculaires qui établissent et contrôlent ce programme chez les vertébrés. Nous nous intéressons également à la manière dont deux processus fondamentaux, la transcription et la réplication, peuvent se dérouler simultanément sur la même molécule d’ADN de façon coordonnée. En effet, des conflits entre ces deux machineries peuvent provoquer une instabilité génétique.
Afin d’obtenir une vision globale de la réplication du génome humain, nous développons des approches à haut débit permettant de cartographier les origines de réplication, leur moment d’activation et les régions du génome sujettes à des dommages (Figure 1). Les résultats obtenus par ces analyses génomiques sont ensuite validés à l’aide d’approches génétiques.
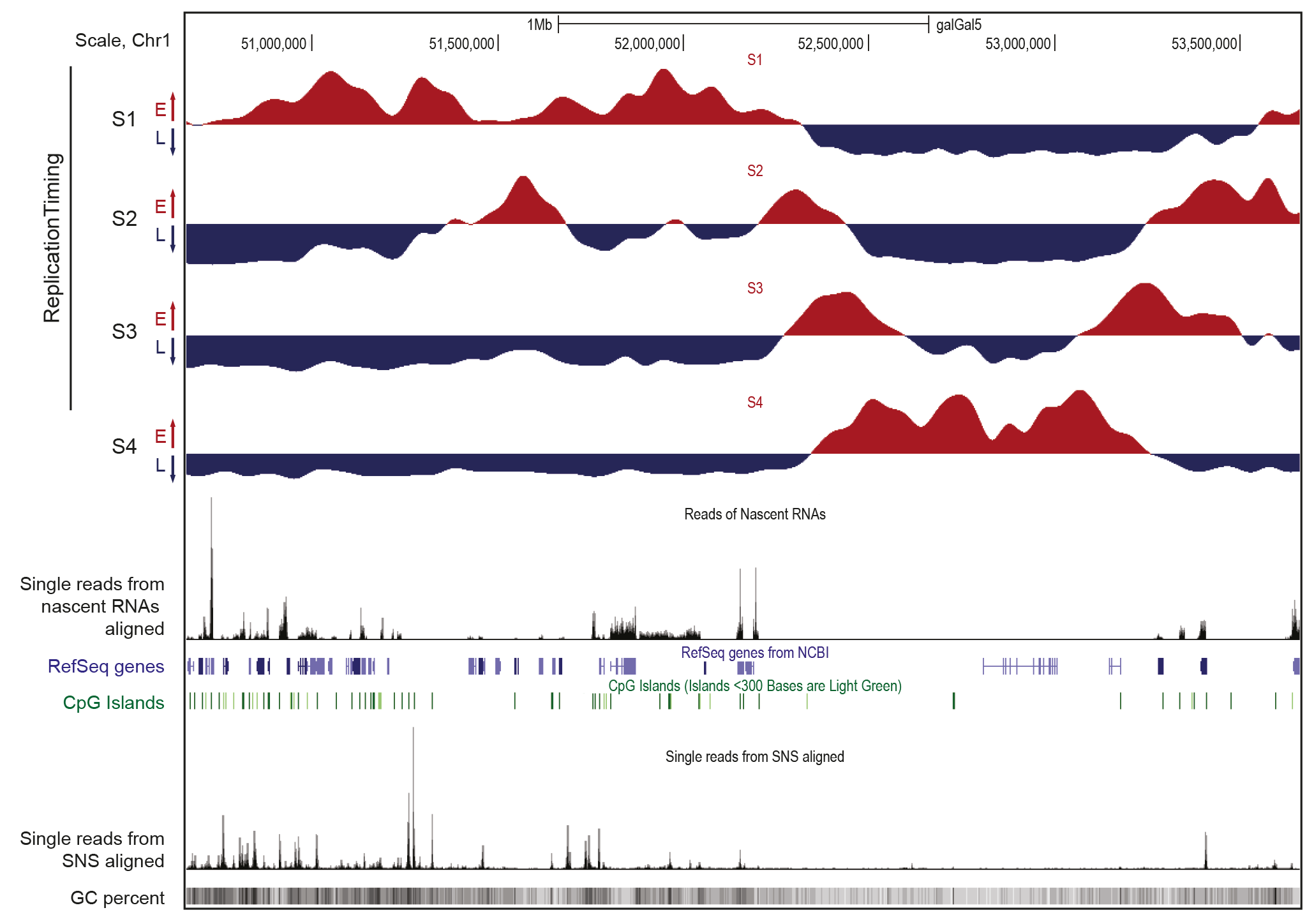
Figure 1. Caractéristiques des domaines de réplication précoce et tardive
Depuis de nombreuses années, nous utilisons un modèle cellulaire aviaire, la lignée lymphoïde DT40, qui se distingue par une recombinaison homologue particulièrement efficace. Ce modèle nous a permis de démontrer que les structures d’ADN appelées G-quadruplex jouent un rôle clé dans l’initiation de la réplication. Nous mettons désormais en place l’utilisation d’une lignée lymphoïde humaine, TK6, que nous modifions génétiquement grâce à l’outil CRISPR/Cas9. Par ailleurs, afin d’étudier l’impact d’un dysfonctionnement de la réplication sur le développement des tissus, nous développons des analyses sur des organoïdes corticaux humains.
Nos projets actuels visent à comprendre comment les G-quadruplex facilitent le recrutement de la machinerie de réplication, ainsi que les interactions réciproques entre les origines de réplication et la transcription.
Comprendre les mécanismes de duplication des génomes eucaryotes est essentiel. La réplication ne garantit pas seulement l’intégrité du génome, elle participe aussi à la coordination des programmes d’expression génique au cours du développement.
Membres
 Theo BARET, PhD student, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Theo BARET, PhD student, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B Caroline DONCARLI, Biology engineer, PRIOLEAU LAB+33 (0)1 57 27 81 24, bureau 520B
Caroline DONCARLI, Biology engineer, PRIOLEAU LAB+33 (0)1 57 27 81 24, bureau 520B William MALLETT, Biology engineer, PRIOLEAU LAB
William MALLETT, Biology engineer, PRIOLEAU LAB Kathrin MARHEINEKE, Researcher, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Kathrin MARHEINEKE, Researcher, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B Aurélie MASSON, PhD student, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Aurélie MASSON, PhD student, PRIOLEAU LAB+33 (0)1 57 27 81 02, bureau 520B
Pour contacter un membre de l’équipe par mail : prenom.nom@ijm.fr
1) Transcription Represses Origin Activity in a Late-Replicating Fragile Site. Mandelbrojt, J., Tonnerre-Doncarli, C., Masson, A., Baret, T., Debatisse, M., and Prioleau, M.-N. (2025). Preprint at bioRxiv, https://doi.org/10.1101/2025.08.01.668051.
2) Dual DNA replication modes: varying fork speeds and initiation rates within the spatial replication program in Xenopus. Ciardo D, Haccard O, de Carli F, Hyrien O, Goldar A, Marheineke K. Nucleic Acids Res. 2025 Jan 24;53(3):gkaf007. doi: 10.1093/nar/gkaf007.
3) Dimeric G-quadruplex motifs-induced NFRs determine strong replication origins in vertebrates.Poulet-Benedetti J, Tonnerre-Doncarli C, Valton AL, Laurent M, Gérard M, Barinova N, Parisis N, Massip F, Picard F, Prioleau MN. Nat Commun. 2023 Aug 10;14(1):4843. doi: 10.1038/s41467-023-40441-4.
4) Polo-like kinase 1 (Plk1) regulates DNA replication origin firing and interacts with Rif1 in Xenopus. Ciardo D, Haccard O, Narassimprakash H, Cornu D, Guerrera IC, Goldar A, Marheineke K. Nucleic Acids Res. 2021 Sep 27;49(17):9851-9869. doi: 10.1093/nar/gkab756.
5) Clustering of strong replicators associated with active promoters is sufficient to establish an early-replicating domain. Brossas C, Valton AL, Venev SV, Chilaka S, Counillon A, Laurent M, Goncalves C, Duriez B, Picard F, Dekker J, Prioleau MN. EMBO J. 2020 Nov 2;39(21):e99520. doi: 10.15252/embj.201899520. Epub 2020 Sep 16. PMID: 32935369
6) Replication dynamics of individual loci in single living cells reveal changes in the degree of replication stochasticity through S phase. Duriez B, Chilaka S, Bercher JF, Hercul E, Prioleau MN. Nucleic Acids Res. 2019 Jun 4;47(10):5155-5169. doi: 10.1093/nar/gkz220. PMID: 30926993
7) Evolution of replication origins in vertebrate genomes: rapid turnover despite selective constraints. Massip F, Laurent M, Brossas C, Fernández-Justel JM, Gómez M, Prioleau MN, Duret L, Picard F. Nucleic Acids Res. 2019 Jun 4;47(10):5114-5125. doi: 10.1093/nar/gkz182. PMID: 30916335
8) Transcription-dependent regulation of replication dynamics modulates genome stability. Blin M, Le Tallec B, Nähse V, Schmidt M, Brossas C, Millot GA, Prioleau MN, Debatisse M. Nat Struct Mol Biol. 2019 Jan;26(1):58-66. doi: 10.1038/s41594-018-0170-1. Epub 2018 Dec 31. PMID: 30598553
9) The spatiotemporal program of DNA replication is associated with specific combinations of chromatin marks in human cells. Picard F, Cadoret JC, Audit B, Arneodo A, Alberti A, Battail C, Duret L, Prioleau MN. PLoS Genet. 2014 May 1;10(5):e1004282. doi: 10.1371/journal.pgen.1004282. eCollection 2014 May. PMID: 24785686
10) G4 motifs affect origin positioning and efficiency in two vertebrate replicators. Valton AL, Hassan-Zadeh V, Lema I, Boggetto N, Alberti P, Saintomé C, Riou JF, Prioleau MN. EMBO J. 2014 Apr 1;33(7):732-46. doi: 10.1002/embj.201387506. Epub 2014 Feb 12. PMID: 24521668
Preprint
Publications
- « Rôle de l’insulateur 5’HS4 du poulet dans la régulation de la réplication » soutenue en Juin 2009 par Vahideh Hassan-Zadeh.
- « Mécanismes moléculaires de l’initiation de la réplication » soutenue en Décembre 2010 par Françoise Meisch.
- « Identification de séquences cis-nucléiques nécessaires à l’initiation de la réplication chez les vertébrés » soutenue en Juin 2014 par Anne-Laure Valton.
- « Construction d’un domaine synthétique de réplication précoce et impact sur la structure chromatinienne et la permissivité transcriptionnelle » soutenue en Septembre 2015 par Caroline Tonnerre-Doncarli Brossas.
- « Mécanismes moléculaires impliqués dans la régulation du moment de déclenchement des origines de réplication » soutenue en Novembre 2016 par Antonin Counillon.
- « Rôle des G-quadruplexes dans la spécification des origines de réplication chez les vertébrés » soutenue en Septembre 2016 par Marc Laurent.
- « Etude moléculaire des éléments cis-régulateurs et de l’organisation de la chromatine des origines de réplication » soutenue en Septembre 2020 par Jérémy Poulet-Benedetti.
« Comment la transcription et le contexte chromosomique interfère avec une origine de réplication efficace ? » soutenue en décembre 2024 par Juliette Mandelbrojt
Nationales :
- Imagerie cellulaire et cytométrie en flux : Plateforme Imago-Seine, Institut Jacques Monod
- Organoïdes corticaux : Plateforme enSCORE, Labex Who am I ?
- Bio-informatique et bio-statistique : LBBE, UCB Lyon 1 (Laurent Duret) et ENS de Lyon (Franck Picard)
- G-quadruplex : Inserm U565, CNRS UMR 7196, MNHN, Paris, France (Patrizia Alberti, Carole Saintomé et Jean-François Riou)
- Sites Fragiles Communs : : Institut Gustave Roussy, Villejuif (Michelle Debatisse et Stéphane Koundrioukoff)
Internationales :
- Organisation tri-dimensionnelle du noyau : University of Massachusetts Medical School, Worcester, Etats-Unis (Anne-Laure Valton et Job Dekker)
- Rôle des G4s dans le positionnement des nucléosomes : Fox Chase Cancer center, Philadelphie, Etats-Unis et Moscow State University, Moscow, Russie (Vasily Studitsky).


